首页 > 医疗资讯/ 正文
人类肠道居住着数以万亿计的微生物,其代谢活动与人体健康密切相关。为精确解析肠道微生物生态系统的分类和功能多样性,构建全面的参考基因组与基因目录至关重要。近年来,统一人类胃肠道基因组(UHGG)联盟等项目整合了多项国际测序数据,成功构建了非冗余的人类肠道微生物参考基因组数据集,但现有参考数据库存在明显地理偏差,欧美人群数据占比超70%,以中国为代表的亚洲人群占比不足30%。
尽管已有研究尝试扩充中国人群微生物参考基因组库,但样本量仍不足以全面揭示中国肠道微生物群的复杂性,这既阻碍了中西方人群的稳健对比分析,更限制了对关键公共卫生领域功能的深入探索。
近日,南京医科大学陈连民、孔祥清团队与复旦大学郑琰、江南大学翟齐啸团队合作,通过整合478,588个高质量微生物基因组,构建了全面的人类肠道微生物组参考数据集(GMR)。该数据集涵盖6,664个物种,其中26.4%为新分类物种,编码超2,000万种独特蛋白质,且47%的蛋白质功能未知。研究团队揭示了中国与非中国人群在物种组成、功能基因及抗生素耐药基因(ARG)方面的显著差异;并将新物种标记基因整合至主流分类数据库,提升了物种检测灵敏度。总之,GMR数据库显著拓展了全球肠道微生物组的组成与功能图谱,为解析种群特异性微生物-宿主互作机制及区域健康差异提供了重要资源。

研究团队收集了来自中国30个省份的6,657个宏基因组样本,通过单样本组装策略成功获得了180,719个宏基因组组装基因组(MAG);然后将新组装的基因组与来自UHGG、hGMB、CGR2和IMGG等现有数据库中的基因组进行整合,经严格的质量控制,最终构建了包含478,588个高质量基因组的GMR数据库,其中247,134个(51.6%)来自中国人群,231,454个(48.4%)来自非中国人群。
评估结果显示,GMR中的基因组质量普遍较高。中国人群来源基因组的中位完整性为90.27%,中位污染率仅为0.88%;非中国来源基因组的中位完整性为90.60%,污染率为0.85%。此外,中国来源基因组的平均大小和N50长度均略高于非中国人群来源基因组。
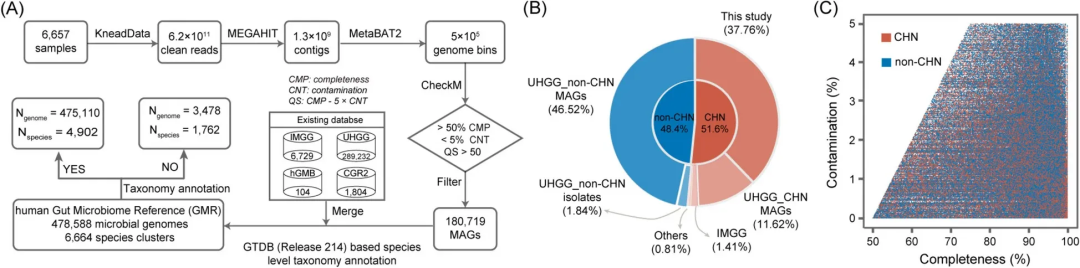
图1. GMR数据库
研究团队利用 GTDB数据库与dRep工具对GMR数据集中微生物基因组进行分类注释。结果显示,共鉴定出6,664个物种级簇,包括6,624个细菌物种和40个古菌物种;其中,1,762个为潜在新物种,微小细菌门(Patescibacteria)的新物种占比最高(43.48%)。
基于GMR数据库的物种分布分析显示,中国人群肠道中检测到4,501个物种,非中国人群为4,308个,两者共享2,145个核心物种;其中,896个核心物种存在显著群体特异性富集。此外,中国人群有2,356个特有物种,主要隶属于芽单胞菌门、酸杆菌门、厚壁菌门D等7个细菌门;而蛭弧菌门未在中国人群基因组中检出,这可能与环境、饮食结构等相关。
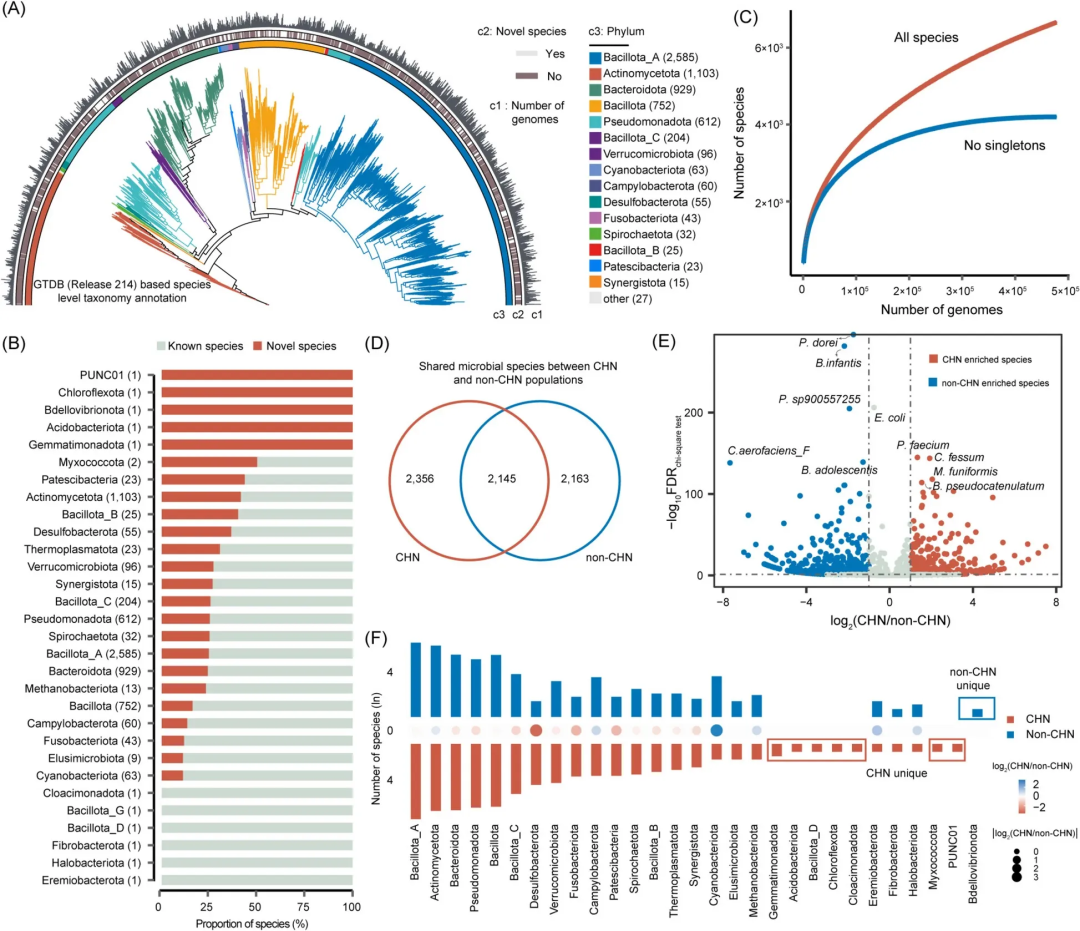
图2. GMR数据库的分类学分类
研究团队对GMR数据库中所有基因组进行全面蛋白编码序列(CDS)预测,共检测到10.28亿个推定基因,平均每个基因组含2,148个CDS;通过多数据库联合注释,发现仅47%的基因能与现有数据库匹配,近半数基因功能未知,热原体菌门的未知功能基因占比最高。
接下来,研究团队分别以50%、90%和100%氨基酸一致性为阈值进行基因聚类分析,最终得到700万-2亿个蛋白质集群(GMRP50、GMRP90和GMRP100)。 GMRP90比较分析显示,中国与非中国人群存在共享及特有蛋白质集群,其中共享649.1万个、中国人群特有656万个、非中国人群特有7,26.2万个。COG功能分布显示,细胞壁/膜生物发生、转录和复制/重组/修复是核心功能类别。
研究团队还分析了抗生素耐药基因(ARG)的分布差异。在51,995个微生物基因组中共检测到1,050个ARG(33.5%),涉及2,233个物种;大肠杆菌、肺炎克雷伯菌、多雷拟普雷沃菌等5个物种携带ARGs数量最多。此外,非中国人群ARGs检出率略高于中国人群,且存在群体特异性富集和特有耐药基因,195个ARGs为中国人群特有,311个ARGs为非中国人群特有;四环素类(Tet)耐药基因的检出频率最高。
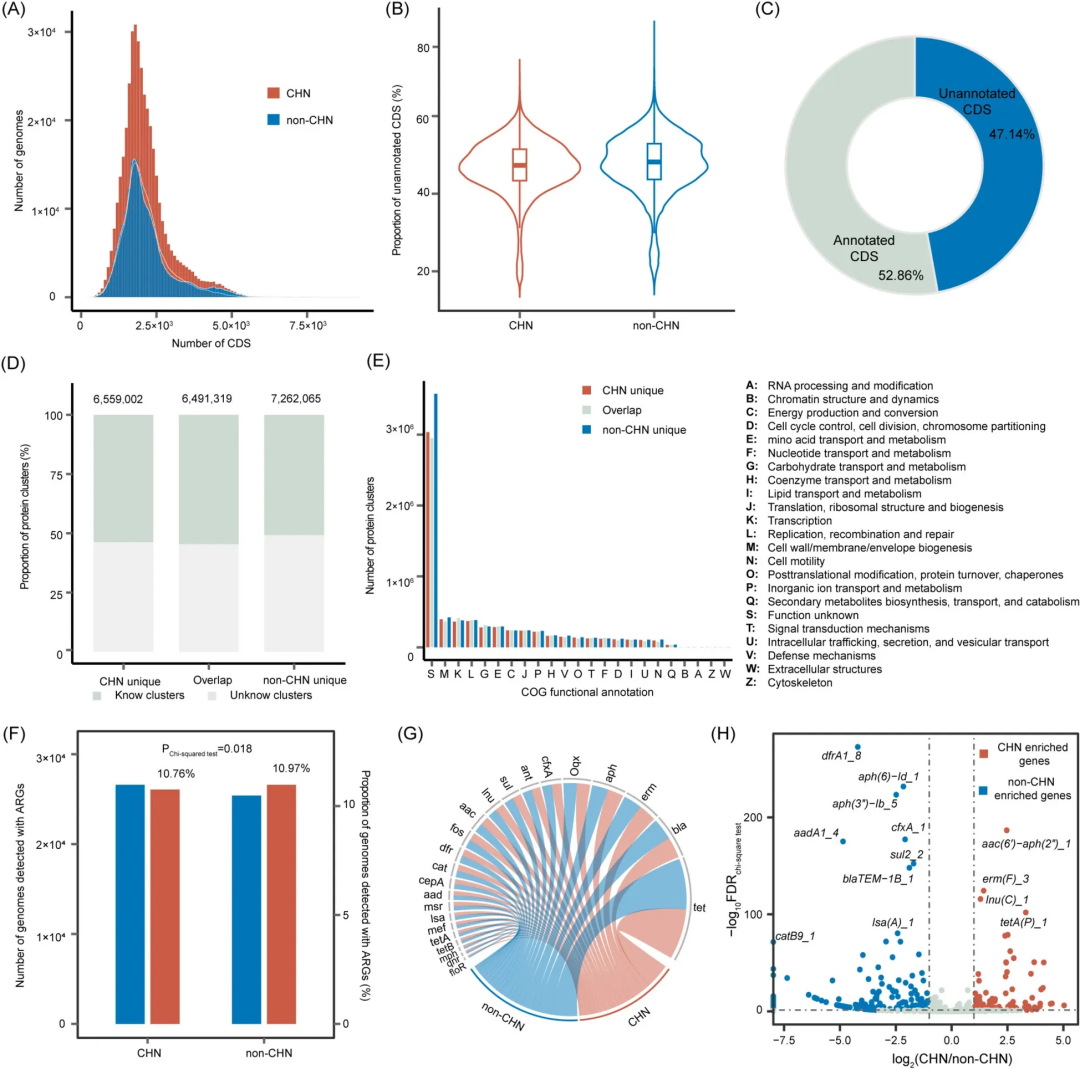
图3. 人类肠道微生物群的功能分类
研究团队对共享的2,145个物种进行深入分析,特别聚焦于304个具有充足基因组数据的物种。通过泛基因组分析和t-SNE降维技术,发现225个物种(74%)存在显著人群特异性遗传结构差异,这种差异在毛螺菌科、拟杆菌科和瘤胃菌科等尤为明显。
以已知具有人群分化特征的普雷沃氏菌属 Copri亚种为参照,研究团队进一步确认了多种菌株(如直肠真杆菌、eligens_A乳球菌)存在类似的遗传分层现象。以eligens_A乳球菌为例,该物种包含4,818个基因组,其遗传结构在t-SNE图上清晰地分为中国和非中国来源两个簇;并通过关联分析发现了4,044个驱动遗传分化的基因,功能涵盖糖脂代谢、抗生素耐药性等。
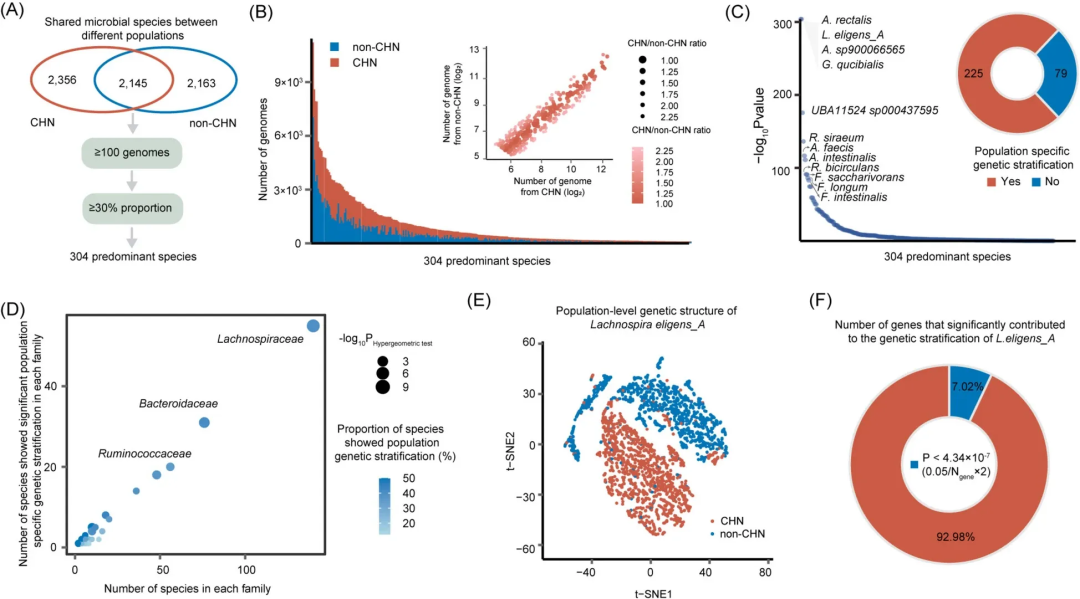
图4. 种群之间优势物种的遗传分层
为提升新发现物种在群体研究中的检出能力,研究团队将GMR数据集的基因组整合至MetaPhIAn4分类数据库,经筛选最终整合了579个非重叠物种的96,156个标记基因,构建了定制数据库。中国、德国、刚果三国独立队列验证显示,该定制数据库展现出卓越的性能。在中国队列,该数据库使样本平均序列比对率提升1%,新增326个物种,物种检出总数提升22.8%;在欧洲和非洲队列中,该数据库分别贡献了8.79%和13.41%物种检出量,证明其具有全球适用性。
利用该定制数据库,研究团队重新分析了包含3,234名中国参与者的CGMR(中国肠道微生物参考)队列,检测到5,310个物种,其中465个为新增物种,97.8%的个体检测到至少1个新增物种。进一步分析发现,新增物种中有54个与22种表型形成325个显著关联,在排除混杂因素后,确定39个物种与16种表型的64个稳健关联,涉及城乡居住环境、年龄、生活方式及饮食习惯等。
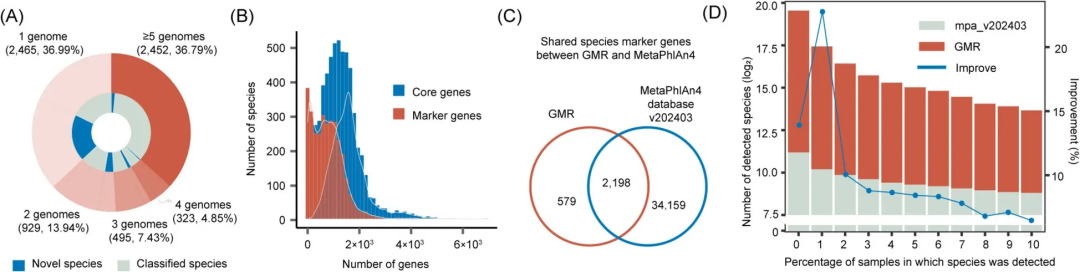
图5. 定制GMR数据库
综上所述,研究团队构建了全球代表性强、人群分布均衡的肠道微生物基因组参考数据库GMR,其揭示的种群特异性物种、遗传分层及功能特征,为解释区域健康差异提供了微生物视角;通过整合该数据集与现有MetaPhlAn4,显著提升了微生物组分析的准确性。GMR数据库将助力肠道微生物组与疾病机制的深入研究,可推动个性化医疗与公共卫生干预的精准化发展。
参考文献:
https://genomemedicine.biomedcentral.com/articles/10.1186/s13073-025-01566-x
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)