首页 > 医疗资讯/ 正文
东亚人群中仍有相当一部分与肺癌相关的遗传元件尚未确定,这凸显了开展大规模全基因组研究的必要性,尤其是在非编码调控方面。
2025年8月9日,浙江大学吴息凤和中国科学院吴逵共同通讯在Nature Communications在线发表题为“Non-coding genetic elements of lung cancer identified using whole genome sequencing in 13,722 Chinese”的研究论文。该研究基于全基因组测序 (WGS) 对 13,722 名中国个体进行了全基因组扫描,以识别与肺癌相关的调控元件。
作者通过对现有东亚研究进行荟萃分析,验证了常见变异位点。整合了 297 名中国个体肺组织的基因组转录组参考序列,作者联结了变异与肺癌的关联,并重点关注了包括 TP63 和 DCBLD1 在内的基因。通过实施 STAAR 流程进行罕见变异的聚合分析,作者在非编码调控背景下识别并复制了包括 PARPBP、PLA2G4C 和 RITA1 在内的新基因。采用基于深度学习的方法,揭示了肺癌相关基因的潜在上游调控因子,例如 TP53、MYC、ZEB1 和 NFKB1。这些发现为肺癌病因的非编码调控提供了重要见解,为干预提供了额外的潜在目标。

肺癌是中国乃至全球最常见的癌症,也是主要的死亡原因,严重威胁着人口健康。种系变异在肺癌的发生发展中起着重要作用,其遗传率估计为18%。大规模全基因组关联研究(GWAS)取得了实质性进展,发现了40多个肺癌相关位点。
然而,一方面,这些关联背后的分子机制尚无定论。鉴于大多数与性状相关的变异位于非编码区域,基因表达调控被认为是填补遗传变异与复杂性状之间知识空白的重要途径之一。基因型-组织表达(GTEx)项目为祖先为欧洲人和非洲人的基因组和转录组数据提供了丰富的多组学参考面板,而亚洲人群中类似规模的多组学参考面板数据却十分有限。事实证明,全面的多组学因果元素图谱是揭示变异型肺癌关联“缺失生物学”的理想且必要的工具。
另一方面,全基因组范围内显著的常见变异只能解释肺癌狭义遗传力中有限的一部分,而很大一部分“缺失遗传力”尚无法解释。新兴证据表明,罕见变异可能在大多数复杂性状中发挥着至关重要的作用。目前,通过基于芯片和全外显子组测序 (WES) 的研究,仅发现了数量有限的单核苷酸变异 (SNV)。基于全基因组测序 (WGS) 的扫描可以捕获所有变异,但主要关注点往往集中在假定的功能丧失 (pLoF) 变异上。位于非编码区(覆盖基因组 97% 以上的区域)的罕见变异的作用在很大程度上仍未被探索。因此,大规模的基于 WGS 的研究对于探索罕见变异元素对肺癌的影响至关重要,尤其是在中国人群中。
为了应对上述挑战,作者对13,722名中国个体进行了一项广泛的全基因组测序(WGS)研究,重点关注非编码调控。作者验证了几种常见的变异-性状关联,并通过整合基因组和转录组参考组以及297个正常肺组织样本,进一步将其投射到转录本水平。作者利用STAAR流程,进行了基于稀有变异的基因中心分析和固定/动态滑动窗口分析,以识别肺癌的潜在易感基因。此外,作者还实施了基于深度学习的计算机精细定位方法,以识别可复制的肺癌相关元件和潜在的上游调控因子。
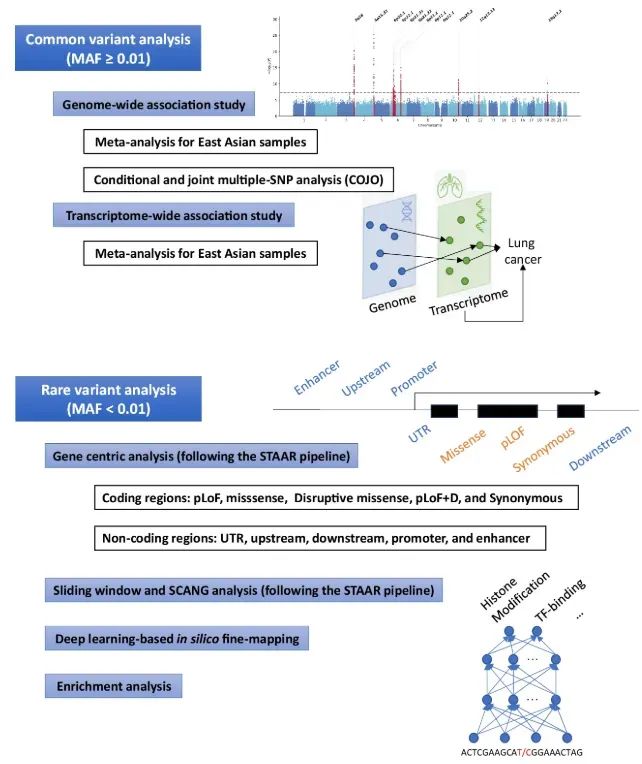
图1 研究概述(图源自Nature Communications)
参考消息:
https://www.nature.com/articles/s41467-025-62459-6
猜你喜欢
- 金枪鱼沙拉的做法-咸鲜味其它工艺菜谱
- 女性保健与养生 什么时候喝水最好
- 娱乐圈美手大赏!王凯、易烊千玺、蔡徐坤谁将胜出?
- 绿色智能家电消费再迎利好
- Molecular Neurodegeneration:中国学者揭示中脑星形胶质细胞源性神经营养因子(MANF)在阿尔茨海默病中的过表达导致突触损失
- 药妆面膜的危害有哪些?可以天天使用吗?
- 【AASLD2024速递】临床治愈患者的肝内整合HBV DNA和转录水平极低
- 杨运桂/张世华团队开发整合组织学图像和空间基因表达谱的深度学习方法STASCAN,破译高分辨率细胞分布
- 通过无创通气降低 PCO2 与改善慢性高碳酸血症呼吸衰竭患者的生存率相关
- 国家卫健委:10月11日新增新冠肺炎本土确诊病例374例本土无症状感染者1386例
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)