首页 > 医疗资讯/ 正文
罕见疾病影响着全球数百万人,但由于患者数据稀缺以及严格的隐私法规如《通用数据保护条例》(GDPR)的限制,其基因组基础仍未被充分理解,这些限制阻碍了跨机构协作分析基因组数据以揭示致病变异的能力。为了应对这一挑战,本研究提出了PRISM,一种基于全同态加密(FHE)的新型隐私保护框架,旨在促进跨多个机构的罕见病变异分析,而无需暴露敏感的基因组信息。PRISM建立在阈值FHE方案之上,通过分散密钥管理来消除单点信任问题,确保没有任何单一实体能够单独解密敏感数据。该方法能够在加密数据上完全执行隐性、显性和新生三种遗传模式下的致病变异过滤,为罕见病研究提供了安全且高效的解决方案。

在方法设计上,PRISM采用BFV-RNS全同态加密方案对患者基因组变异数据进行加密处理,数据以VCF文件形式存储,并通过数值编码将基因型转换为整数形式以便进行同态运算。针对每种遗传模型,研究提出了两种算法变体:乘法密集型(MUL-IN)和加法密集型(ADD-IN)。MUL-IN方法依赖于大量乘法操作进行变异与查询的逐样本比较,并通过乘法聚合结果,其计算逻辑严密但运行成本较高。ADD-IN方法则通过减少乘法操作、增加加法操作来显著提升效率,尽管会生成更大的密文,但通过SIMD(单指令多数据)和多重线程并行处理,仍能有效处理数百万规模的变异数据。具体而言,在隐性/显性模型中,ADD-IN通过计算样本比较结果的加和并与总样本数相减,再引入随机加密值与乱序操作以保护隐私;在新生模型中,该方法分别处理患者组与未患病组的变异存在与否的加和,并通过类似策略实现安全过滤。
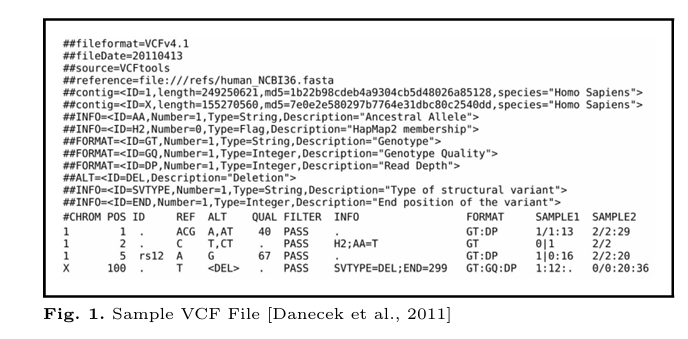
图1 VCF文件
实验部分在Google Cloud高性能实例上展开,使用真实罕见病数据(6例颅面胸发育不良患者外显子数据)和模拟数据对不同算法和已有方案进行性能对比。结果显示,ADD-IN方法在隐性/显性模型过滤中相比MUL-IN最高可实现17倍加速,在新生模型中最高可达22倍。例如,针对128个样本、160万个变异的数据,ADD-IN仅需15秒,而MUL-IN需4分14秒。
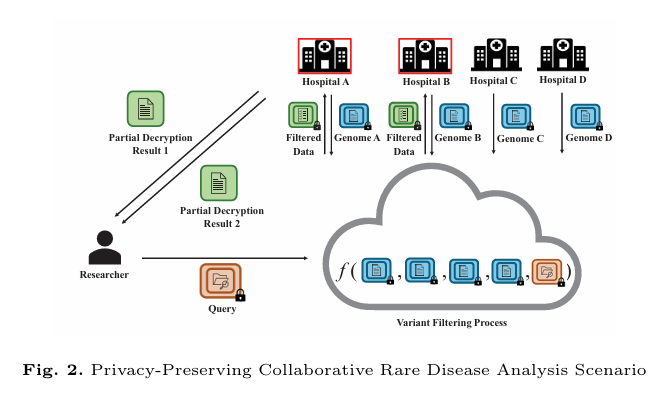
图2 隐私保护协作罕见病分析场景
与基于安全多方计算(MPC)的现有方案[Akgun et al., 2020]相比,PRISM在样本数不超过128时表现相当,且由于采用单云模型避免了多方服务器间的通信延迟与信任假设问题,但其计算开销随数据规模增长而增加。
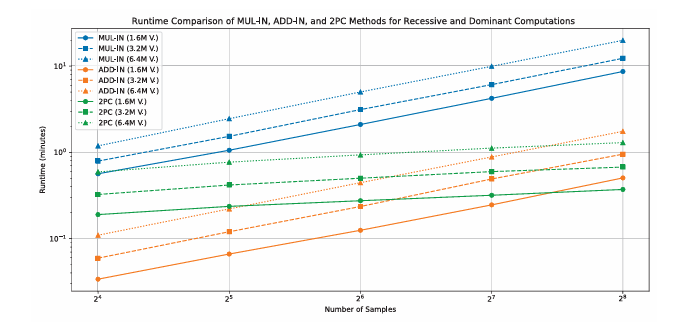
图3 MUL-IN、ADD-IN和2PC方法在隐性和显性计算中的运行时间比较
实验还表明,通过256线程并行,ADD-IN可处理高达4096样本、640万个变异的数据,耗时在数分钟内,体现了实际应用的可行性。
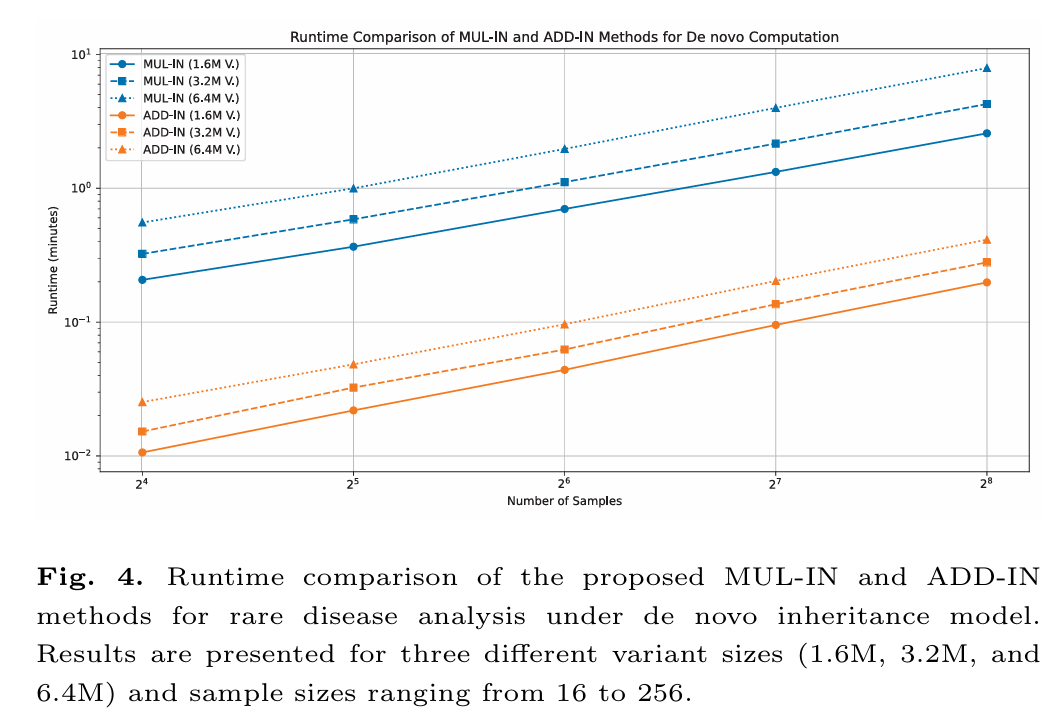
图4 MUL-IN和ADD-IN方法在新生计算中的运行时间比较
在安全模型方面,PRISM采用分布式密钥生成与阈值解密机制,密钥管理委员会由多个机构组成,任何单一机构无法独立完成解密,系统假设不超过设定阈度的委员会成员合谋,云服务器被视为“诚实但好奇”,研究者则被限制只能访问经审核的查询结果。为进一步防止信息泄露,系统实施查询审核与捆绑策略,要求研究者提交大规模分析请求(如全基因组或外显子组级别),避免针对特定变异或个体的探测,同时通过自动化脚本过滤已知与研究无关的致病变异,保护患者“不知情权”。密文输出方面,MUL-IN仅返回加密的匹配变异计数,ADD-IN则返回标记匹配位置的乱序加密向量,两者均能有效保护原始基因型数据不被泄露。
原始出处:
Akkaya, G., Erdogmus, N., & Akgun, M. (2025). PRISM: Privacy-preserving Rare Disease Analysis using Fully Homomorphic Encryption. Bioinformatics, btaf468. https://doi.org/10.1093/bioinformatics/btaf468
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)