首页 > 医疗资讯/ 正文
近年来,基因组学技术的快速发展为罕见病的诊断带来了革命性的突破,尤其是针对超罕见单基因疾病的研究。传统的诊断方法依赖于家系分离分析或小规模队列研究,但对于表型高度异质且病例稀少的疾病,这些方法往往难以奏效。随着国际罕见病队列规模的扩大,联合分析跨队列数据成为可能,但如何开发校准良好的统计遗传学方法仍是当前研究的重点。美国未确诊疾病网络(UDN)通过整合临床、研究和实验中心资源,为基于病例的诊断分析提供了规模化平台。本研究首次对UDN患者的全基因组测序数据进行了联合分析,并开发了新的统计方法,用于优先考虑新生突变和复合杂合突变的疾病基因,同时检测了候选基因和已知诊断基因的富集通路。研究还发布了自动化分析工具RaMeDiES,支持跨队列的去标识化数据分析,为罕见病诊断和研究提供了新思路。

研究团队首先对UDN队列的4236名个体进行了全基因组测序数据的统一处理和变异调用,确保数据质量。通过开发Rare Mendelian Disease Enrichment Statistics(RaMeDiES)软件包,团队实现了对新生突变(de novo)和复合杂合突变(compound heterozygous)的统计优先级分析。RaMeDiES-DN方法专注于新生突变的富集检测,通过整合单核苷酸变异(SNV)和插入缺失(indel)的突变率模型以及有害性预测评分,显著提高了统计效能。
该方法利用变异特异性突变率和功能评分,计算基因水平的突变靶标,并通过解析公式评估累积概率,避免了传统置换检验的计算负担。此外,研究还扩展了RaMeDiES-DN的应用范围,纳入深度内含子剪接变异,利用SpliceAI评分和体外大规模平行剪接报告实验(MPSA)验证其功能影响。对于复合杂合突变,团队开发了RaMeDiES-CH方法,通过条件概率模型避免了对群体结构的直接依赖,并利用突变率模型评估变异对的空间分布。为进一步提高诊断率,研究还进行了通路富集分析,通过表型相似性聚类和基因集富集分析(GSEA)识别潜在的功能单元。
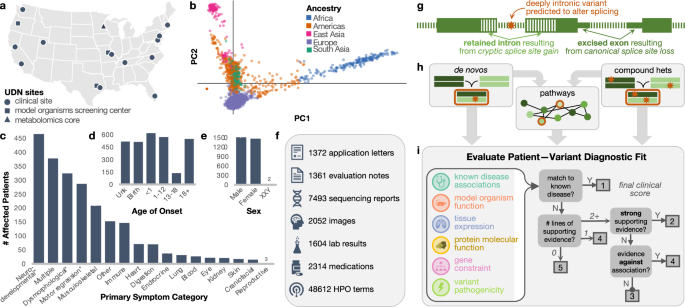
图1:未确诊疾病网络队列分析
结果显示,RaMeDiES-DN在UDN队列中成功识别了多个显著富集的新生突变基因。例如,KIF21A基因的新生突变与一名患者的完全诊断和另一名患者的部分诊断高度相关。通过整合GeneBayes值(评估基因对杂合截断变异的负选择强度),研究进一步发现了BAPI和RHOA等基因的致病性变异,这些基因在患者中表现出强烈的临床匹配性。
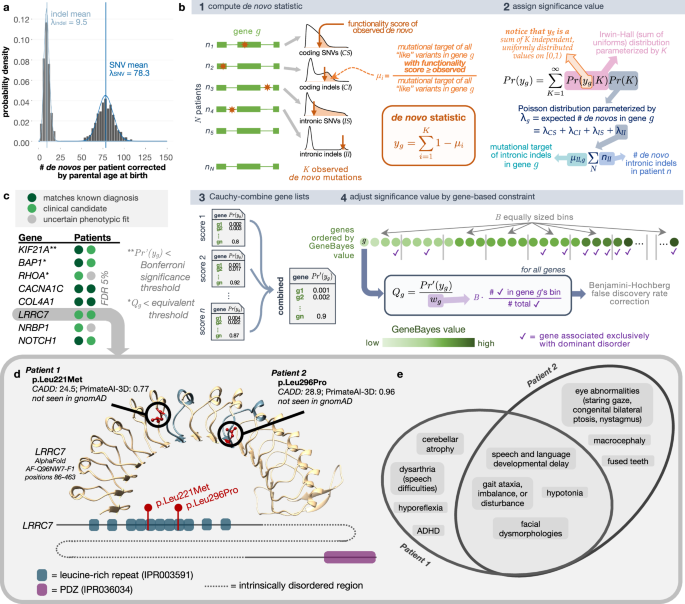
图2:新生突变统计计算及基因发现
此外,团队还鉴定出LRRC7基因的新生突变与神经发育障碍的关联,相关病例已被纳入临床病例系列研究。在纳入非外显子剪接变异后,尽管未发现新的显著基因,但该方法为全基因组数据的全面分析提供了可行框架。与现有方法DeNovoWEST相比,RaMeDiES-DN在保持相似检出率的同时,显著提高了计算效率。
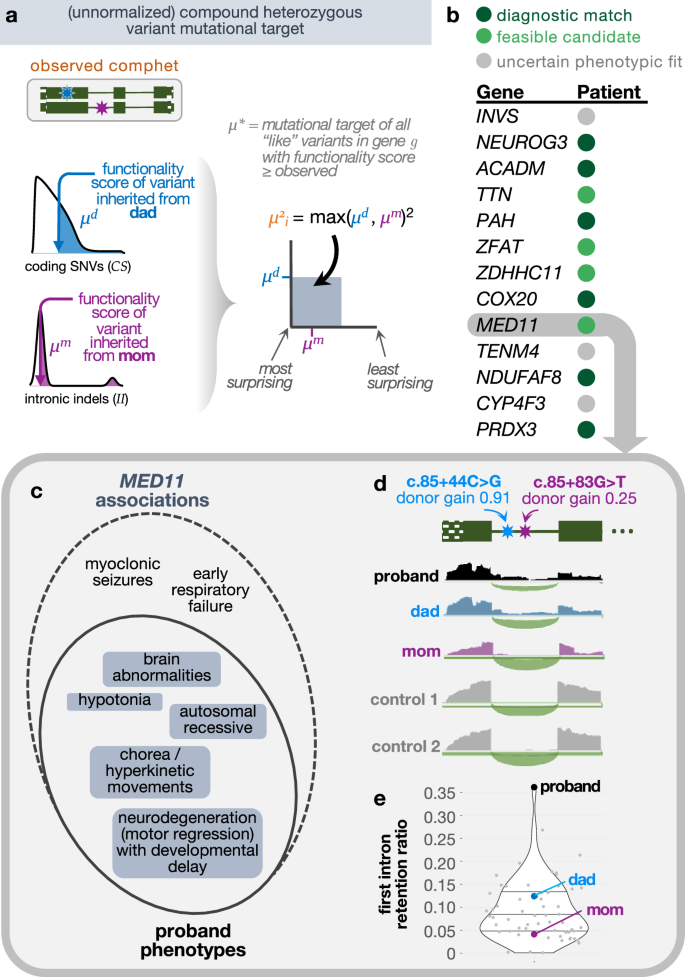
图3:复合杂合变异分析
对于复合杂合突变,RaMeDiES-CH虽未发现显著的基因富集,但个体水平的分析(RaMeDiES-IND)成功识别了多个已知诊断基因(如NEUROG3、PAH等)和新的候选基因(如MED11)。MED11的深内含子变异通过RNA测序验证了其剪接异常效应,为一名神经退行性疾病患者提供了潜在诊断依据。通路分析则揭示了免疫蛋白酶体复合物和跨膜信号通路在特定患者亚群中的富集,为疾病机制和靶向治疗提供了线索。
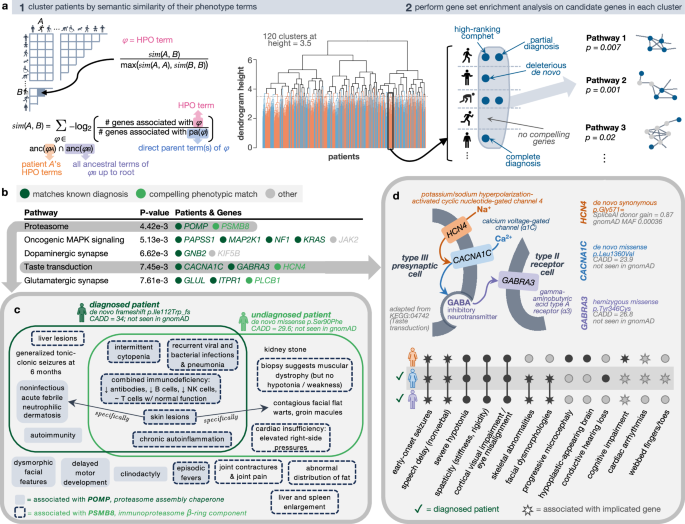
图4:表型相似患者亚群中富集的生物通路
综上所述,本研究通过开发校准良好的统计方法和多维度分析框架,显著提升了超罕见单基因疾病的诊断能力。RaMeDiES工具的开源发布和公共数据浏览器的建立,为国际罕见病研究的协作与数据共享奠定了基础。未来,随着测序技术的进步和队列规模的扩大,联合基因组分析有望成为罕见病诊断的核心手段,为全球患者带来更高效的精准医疗服务。
原始出处:
Nadimpalli Kobren, S., Moldovan, M.A., Reimers, R. et al. Joint, multifaceted genomic analysis enables diagnosis of diverse, ultra-rare monogenic presentations. Nat Commun 16, 7267 (2025). https://doi.org/10.1038/s41467-025-61712-2
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)