首页 > 医疗资讯/ 正文
[摘要]背景与目的:胃癌作为常见恶性肿瘤之一,早期诊断和治疗对改善患者预后至关重要。本研究基于早期胃癌患者的血清定量蛋白质组学研究,旨在发现潜在诊断早期胃癌的生物标志物。方法:收集2023年6月—12月在兰州大学第二医院就诊的原发性胃癌患者血清的临床资料与体检的健康对照人群的血清资料。所有资料根据本研究的纳入和排除标准进行筛选。使用数据依赖性的采集模式(data-dependent acquisition,DDA)建立蛋白质谱图库后,数据非依赖性的扫描模式(data-independent acquisition,DIA)对各样本进行单独分析。通过STRING数据库分析蛋白组学中胃癌血清中表达上调的蛋白质相互作用,采用KEGG与基因本体GO数据库分析蛋白质对应基因的信号转导通路与功能注释。通过GEPIA2分析基因在胃癌与非胃癌组织中的表达水平,Kaplan-Meier Plotter分析各基因对应胃癌患者的总生存期。利用定量反转录聚合酶链反应(quantitative reverse transcription polymerase chain reaction,qRT-PCR)法验证差异基因在临床胃癌及癌旁组织中的表达。本研究获得兰州大学第二医院研究伦理委员会的批准(伦理批号:2023A-459)并豁免知情同意。结果:最终入组并获得30例原发性胃癌患者血清与29例健康对照人群血清资料,另获得8例癌旁组织资料。采用血清定量蛋白质组学共鉴定到666种交集蛋白,其中在胃癌组中有16种蛋白质对应基因表达上调,22种表达下调(P<0.05,|FC|≥1.5)。 STRING数据库分析显示,10种上调蛋白质处于互作网络中,KEGG与GO分析表明基因与癌症发生、发展的生物过程密切相关。GEPIA 2与Kaplan-Meier Plotter分析显示B2M、TAGLN2、CTSD、HSP90AB1、SH3BGRL3与CFL1共计6个基因在胃癌组中高表达(P<0.05)且预后不良。qRT-PCR结果表明,TAGLN2、CTSD、SH3BGRL3、CFL1与HSP90AB1在胃癌组织中高表达(P <0.05)。结论:TAGLN2、CTSD、SH3BGRL3、CFL1与HSP90AB1具有作为诊断早期胃癌的血清生物标志物的潜力,有利于胃癌的早诊早治。
[关键词] 蛋白质组学;胃癌;生物标志物;早期诊断
[Abstract] Background and purpose: Gastric cancer, as one of the most common malignant tumors, requires early diagnosis and treatment to improve patient prognosis. This study, based on serum quantitative proteomics research of early-stage gastric cancer patients and non-gastric cancer patients, aims to identify potential diagnostic biomarkers for early gastric cancer. Methods: Serum samples from primary gastric cancer patients and healthy control individuals were collected from Lanzhou University Second Hospital between June and December 2023, following inclusion and exclusion criteria. A protein spectral library was established using Data-Dependent Acquisition (DDA) mode, and each sample was analyzed using Data-Independent Acquisition (DIA) mode. The STRING database was used to analyze protein-protein interactions of upregulated proteins in gastric cancer serum. Kyoto Encyclopedia of Genes and Genomes (KEGG) and Gene Ontology (GO) were used to analyze the pathways and functional annotations of the corresponding genes. Gene expression levels in gastric cancer and non-gastric cancer tissues were analyzed using GEPIA 2, and overall survival of each gene in gastric cancer was analyzed using Kaplan-Meier Plotter. Differential gene expression in clinical gastric cancer and adjacent tissues was validated by quantitative reverse transcription polymerase chain reaction (qRT-PCR) This study was approved by the Ethics Committee of Lanzhou University Second Hospital (Ethical No.: 2023A-459) and was exempt from the informed consent. Results: Finally, serum samples from 30 primary gastric cancer patients, 29 healthy control individuals, along with the para-cancerous tissues from 8 patients were collected. A total of 666 intersecting proteins were identified through serum quantitative proteomics. Among them, 16 proteins showed upregulated expression and 22 proteins showed downregulated expression in the gastric cancer group (P<0.05, |FC|≥1.5). STRING database analysis showed that 10 upregulated proteins were involved in interaction networks. KEGG and GO analysis indicated that these genes were closely related to the biological processes of cancer occurrence and development. GEPIA 2 and Kaplan-Meier Plotter analysis showed that 6 genes, B2M, TAGLN2, CTSD, HSP90AB1, SH3BGRL3, and CFL1, which were highly expressed in the gastric cancer group (P<0.05) and associated with poor prognosis. Clinical verification by qRT-PCR confirmed that TAGLN2, CTSD, SH3BGRL3, CFL1 and HSP90AB1 were highly expressed in gastric cancer tissues (P<0.05). Conclusion:TAGLN2, CTSD, SH3BGRL3, CFL1, and HSP90AB1 have the potential to serve as clinical early gastric cancer diagnostic serum biomarkers, which may facilitate early diagnosis and treatment of gastric cancer.
[Key words] Proteomics; Gastric cancer; Biomarkers; Early diagnosis
胃癌是常见的恶性肿瘤之一,其早期诊断和治疗对改善患者预后至关重要。2020年,胃癌在全球癌症发病率中排名第5,在癌症相关死亡率中排名第4[1]。虽然在过去30年中其年龄标准化发病率和死亡率有所下降,但预计到2040年,胃癌的全球负担将增加62%[1-2]。
包括癌胚抗原(carcinoembryonic antigen,CEA)、甲胎蛋白(alpha fetoprotein,AFP)、糖类抗原19-9(carbohydrate antigen 19-9,CA19-9)和CA72-4在内的几种肿瘤标志物已被广泛用于胃癌的临床血清学诊断,然而它们的敏感性和特异性并不理想[3]。肿瘤免疫疗法和生物标志物已成为增强诊断和治疗决策的有前途的策略。有研究[4-5]从肿瘤微环境(tumor mircroenvironment,TME)和免疫浸润的角度确定了胃癌生物标志物,主要依赖于癌症基因组图谱(The Cancer Genome Atlas,TCGA)和基因表达总库(Gene Expression Omnibus,GEO)等数据集。然而,这些研究并未深入探索临床血清样本中的差异表达基因。此外,循环肿瘤细胞和循环非编码RNA等新型生物标志物比传统血清肿瘤标志物显示出更高的特异性,但样本获取不如血清便利,很难在临床上广泛开展[3]。
本研究基于定量蛋白质组学鉴定了早期胃癌患者和非胃癌人群对照的血清中差异表达的蛋白质。利用STRING、GEPIA 2和Kaplan-Meier Plotter数据库对这些差异表达基因进行进一步分析,并采用定量反转录聚合酶链反应(quantitative reverse transcription polymerase chain reaction,qRT-PCR)法验证胃癌和癌旁组织中的差异表达基因,进而筛选出可望用于早期胃癌诊断的生物标志物。
1 资料和方法
1.1 临床血清和组织样本采集
收集2023年6月—12月在兰州大学第二医院就诊的原发性胃癌患者血清的临床资料(n=1 527)与体检的健康对照人群的血清资料(n=1 386),对所有资料根据本研究的纳入和排除标准进行筛选。本研究的胃癌患者的纳入标准为:① 经组织病理学诊断为原发性胃癌的患者;② 经胃镜下钳取活组织病理学检查诊断为胃癌的患者;③ 年龄≥18岁;④ 进行过CEA、AFP、CA19-9、CA72-4等传统肿瘤标志物检查[6]。排除标准为:① 经胃镜证实存在食管、十二指肠溃疡及其他消化系统病变者;② 临床病理学资料不完整的患者;③ 已经接受药物治疗的胃癌患者; ④ 已经在外院接受手术的胃癌患者;⑤ 存在局部淋巴结侵犯和远处转移的胃癌患者。健康对照人群的排除标准为:① 存在胃良性溃疡、胃良性肿瘤、胃淋巴瘤、胃肠道间质瘤、胃神经内分泌肿瘤等非胃癌消化系统疾病的患者;② 有癌症家族史;③ 存在其他合并感染、血常规检查指标异常的患者;④ 脂血、溶血等血液样本异常的患者。对照组均来自体检的健康人群。肿瘤分期根据国际抗癌联盟(Union for International Cancer Control,UICC)第8版TNM分类系统进行。所有血液样本都收集在含有凝块活化剂的血清分离管中以获得血清。另采集兰州大学第二医院原发性胃癌患者的原发性胃癌组织和癌旁组织。其中癌旁组织指距肿瘤边缘≥3 cm的非癌变胃黏膜组织,经病理学检查确认无肿瘤细胞浸润。本研究将原发性胃癌视为早期胃癌。
本研究获得兰州大学第二医院研究伦理委员会的批准(2023A-459)并豁免知情同意。采集的配对胃癌组织和邻近组织样本用磷酸盐缓冲液(phosphate-buffered saline,PBS)清洗后,使用RNA稳定保存液保存于-20 ℃。PBS与RNA稳定保存液均购自武汉赛维尔生物科技有限公司。
1.2 血清蛋白质组学样本制备与分析
将非转移性胃癌(gastric cancer, non-metastatic,GCNM)组和健康对照组(normal control,NC)各分为3个亚组(GCNM-1/2/3与NC-1/2/3)。每个亚组内的样本都经过汇集、消化、脱盐和分馏。随后将每个亚组的血清混合后进行蛋白质组学检测,以降低个体引起的组间差异。使用Nomimicro/通用蛋白质消化试剂盒(Naomi MagicOmics-MMB8X)消化蛋白质。向含有MMB珠的孔中加入20 μL蛋白溶液,37 ℃温育30 min。然后加入45 μL结合缓冲液,室温下轻轻振荡温育15 min。温育结束后,弃去上清液,用洗涤缓冲液将MMB珠洗涤3次。随后,加入20 μL消化工作液重悬珠子,37 ℃温育至少4 h。加入5 μL的淬灭缓冲液终止反应,然后冻干样品。将标记的样品溶解在100 μL流动相A液中,14 000×g离心20 min,收集上清液,以 0.7 mL/min的流速进行高效液相色谱分离。采用采集模式(data-dependent acquisition,DDA)为GCNM组和NC组中每个亚组的集合样本建立光谱库。制备流动相A液(100.0%水,0.1%甲酸)和B液(80.0%乙腈,0.1%甲酸)。将冻干样品溶解在10 μL流动相A液中,在4 ℃下以14 000×g离心20 min,然后注入400 ng上清液进行分析。DDA分析使用了配备 CaptiveSpray离子源的timsTOF_HT质谱仪,质量范围为300~1 500 m/z。第一质谱分辨率设置为60 000(1 222 m/z),TIMS通道中的累积时间为100 ms。毛细管电压设定为1.6 kV,迁移率范围为0.6~1.6 cm2/(V·s)。总循环时间为1.1 s,PASEF循环10次,分离流速为500 nL/min。按照与DDA相同的初始方案,对每个样品单独进行数据非依赖性的扫描模式(data-independent acquisition,DIA)分析。在DIA模式下,累积时间设定为50 ms,毛细管电压为1.5 kV,迁移率范围为0.70~1.30 cm²/(V·s)。总循环时间为1.23 s,流速为300 nL/min。最后,利用分析软件进行数据整合,得出血清蛋白的定量结果。质谱蛋白质组学数据已通过iProX合作伙伴资源库存入ProteomeXchange Consortium(http://proteomecentral.proteomexchange.org),数据集标识符为PXD061032[7]。我们采用了多种质控方法以确保数据的可靠性。累计回收率评估不同样本间的蛋白鉴定一致性,确保了分析的全面性。分析每个色谱峰的平均数据点数来保证数据的质量,确保足够的数据密度用于定量分析。同时,峰容量用于衡量色谱柱的分离能力,确保高效分离蛋白质。使用假阳性率(false discovery rate, FDR)筛选了蛋白质的定性结果,以确保鉴定结果的高可信度。Nomimicro/通用蛋白质消化试剂盒购自中国青莲百奥生物科技有限公司。
1.3 STRING数据库分析
STRING数据库(https://cn.string-db.org/)用于分析血清蛋白质组学中发现的上调蛋白之间的相互作用[8]。最低交互作用得分设定为0.4,未出现在交互作用网络中的蛋白质被排除在外。选出处于相互作用网络的上调基因进行后续分析。
1.4 GO和KEGG分析
GO分析采用标准化的三级结构:生物过程(biological process,BP)、细胞组分(cellular component,CC)和分子功能(molecular function,MF)来定义和描述基因和蛋白质的功能。KEGG分析用于在分子水平上对基因进行高级功能注释。通过GO和KEGG分析对选定的10个基因进行了功能注释。
1.5 GEPIA 2数据库分析
GEPIA 2数据库(http://gepia2.cancer-pku. cn/)支持对84种癌症亚型的基因和转录水平进行定量基因表达分析,并支持亚型之间的比较[9]。GEPIA 2用于分析胃癌和正常组织之间10种选定蛋白质对应基因的差异表达,阈值设定为 |log2FC|=0.5。
1.6 Kaplan-Meier plotter数据库分析
Kaplan-Meier plotter数据库(https://www. kmplot.com/analysis/)可用于评估21种癌症类型、35 000个样本中胃癌基因表达(mRNA、miRNA、蛋白质和DNA)与总生存期之间的关系[9]。
1.7 定量反转录聚合酶链反应分析
对8例配对的胃癌组织样本和8例癌旁组织样本进行qRT-PCR分析,以验证枢纽基因在组织中的差异表达。从胃癌和邻近非肿瘤组织中提取总RNA。使用DS-11分光光度计(DeNovix DS-11)测定RNA浓度。使用SPARKscript Ⅱ All-in-one RT SuperMix for qPCR对提取的1 μg RNA进行反转录,并在反转录过程中去除基因组DNA。将得到的cDNA稀释5倍,然后2×SYBR Green qPCR混合液将1.8 μL稀释的cDNA用于qPCR检测。Bio-Rad CFX96试剂(美国Bio-Rad公司)用于相对基因表达进行qRT-PCR分析。每个反应的总体积为20 μL,由目的基因引物和SYBR Green预混剂组成。扩增条件包括在95 ℃下初始变性30 s,然后在95 ℃下变性5 s,在60 ℃下退火/延伸30 s,循环40次。使用默认的熔融曲线分析程序确认qRT-PCR结果的特异性。数据归一化采用2-ΔΔCT方法。每个样本检测3次,通过计算平均CT值得出最终结果。此外,用无酶水作为无模板对照以排除污染的可能性。6个基因的引物序列见表1。所有目的基因与内参基因GAPDH引物购自生工生物工程(上海)有限公司。RNA提取FreeZol试剂购自南京诺唯赞生物科技股份有限公司,反转录与qRT-PCR试剂均购自山东思科捷生物技术有限公司。
1.8 数据处理和统计学分析
使用Spectronaut中的Pulsar搜索引擎进行血清蛋白质组学分析,利用DDA和DIA数据创建光谱库,并评估各样本的肽和蛋白质鉴定情况。UniProt数据库(智人:蛋白质数量为20 407;下载日期为2023年3月7日)用于量化每个样本中鉴定出的蛋白质,非空蛋白质可获得量化值。维恩图分析用于确定组内和组间差异,取6个亚组间鉴定出的蛋白质交集。对原始数据进行中值归一化处理,以尽量减少实验偏差,并去除含有零值的行,以获得归一化的蛋白质定量结果。差异倍数(fold change,FC)法用于评估胃癌组相较对照组的蛋白表达差异倍数。对于两组间共有的蛋白质,其对应的FC=胃癌组蛋白丰度/对照组蛋白丰度。对蛋白质差异表达的数据进行正态性检验和方差齐性分析,若符合,则采用独立样本t检验筛选差异表达的蛋白质,否则则采用非参数检验(Wilcoxon秩和检验)。P<0.05和|FC|≥1.5为差异有统计学意义。FC≥1.5认为基因在胃癌组表达上调,FC≤-1.5则认为在胃癌组中表达下调。生物信息学在线分析平台微生信(https://www.bioinformatics.com.cn)[10]用于显示血清蛋白质组学、GO和KEGG分析的结果,结果以折合富集度和-log10P表示。使用 GraphPad Prism 8.0.1 软件(GraphPad Software, Inc., USA)进行统计学分析。

2 结 果
2.1 早期胃癌患者和健康对照的血清蛋白质组学分析
基于纳排标准,本研究最终获得30例GCNM组患者和29例NC组的血清样本(图1)。每组分为3个亚组,各亚组血清混合后分别进行全面的定性和定量蛋白质组学分析,以判定两组人群血清蛋白的差异。在分析前发现1例健康对照血清样本发生溶血且无法分层,遂予以剔除。对样本血清进行血清蛋白质组学分析。质控分析显示累计回收率稳定增长,证明样本的蛋白鉴定完整性良好;平均数据点数表明色谱峰提供了足够的定量数据,确保分析的精确性;峰容量分析结果表明色谱分离效果优良,保证了分析的分辨率;使用FDR方法进行蛋白质定性分析,确保了鉴定结果的可靠性,假阳性率控制在可接受范围内。总体而言,质控结果表明数据质量较高,分析结果可信。血清蛋白质组学的密度雨云图显示了两组血清蛋白的相对丰度,连线表示GCNM组蛋白质的平均相对丰度更高(图2A)。NC组共鉴定出717种蛋白质,其中573种为重叠蛋白质,占79.92%。在GCNM组中,共鉴定出750种蛋白质,其中有597种为重叠蛋白质,占79.60%(图2B)。GCNM组和NC组共鉴定出666种蛋白质,并进行了蛋白质组学分析。热图显示了差异表达基因的组间的聚类和不同的相对丰度特征(图2C)。火山图显示共有38个基因的相对表达差异有统计学意义(|FC|≥1.5,P<0.05),其中GCNM组中有16个基因表达上调,22个基因表达下调(图2D,表2)。
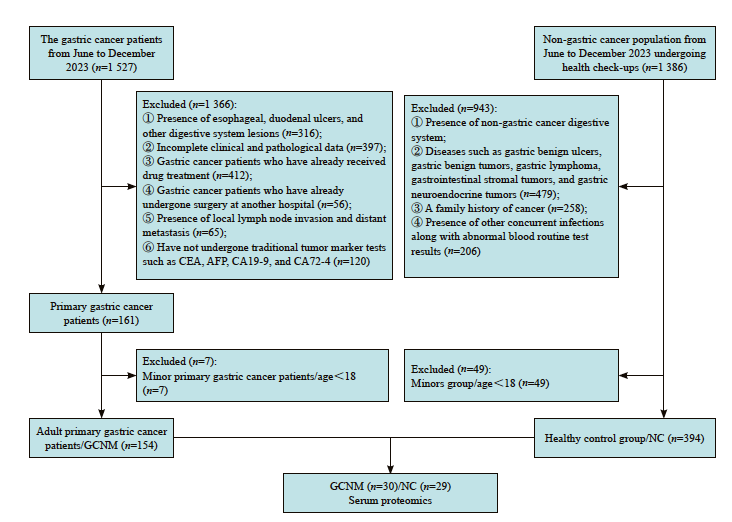
图1 患者及对照人群的纳入和排除标准的流程图
Fig. 1 Flow diagram of participants’ selection
GCNM: Gastric cancer, non-metastatic; NC: Healthy normal control.
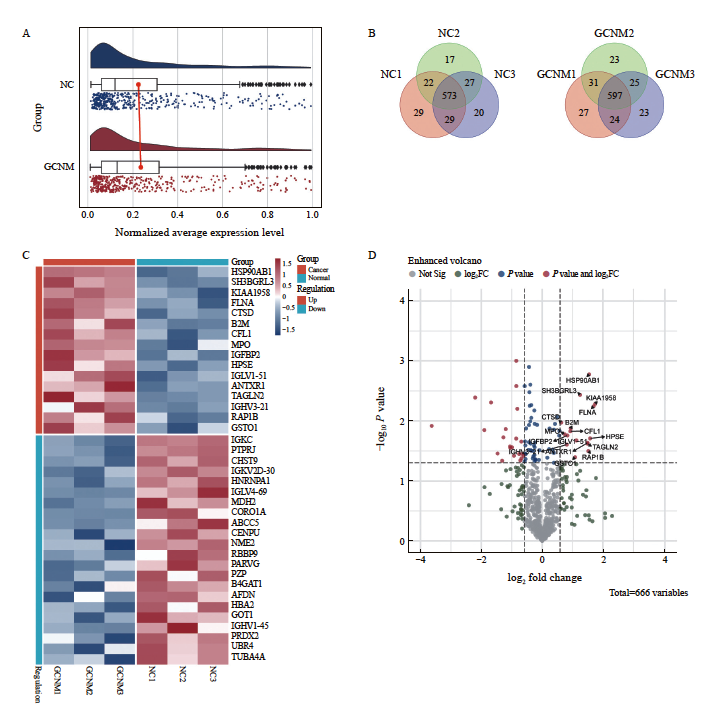
图2 GCNM患者和NC的血清样本的蛋白质组学分析
Fig. 2 Proteomic analysis of serum samples from patients with non-metastatic gastric cancer (GCNM) and healthy normal control (NC) groups
A: Raincloud plot illustrating the normalized relative abundance of serum proteins, summarizing the median, mean, and confidence interval. The X-axis represents the log-transformed normalized relative abundance of each protein. B: Overlap analysis of identified proteins in the GCNM and NC groups. A total of 717 proteins were detected in the NC group, with 573 overlapping proteins (79.92%). In the GCNM group, 750 proteins were identified, including 597 overlapping proteins (79.60%). C: Heatmap comparing the expression of all differentially expressed proteins across different groups, where red indicated high expression and blue indicated low expression. D: Volcano plot illustrating gene expression differences between the GCNM and NC groups. Each dot representd an individual protein. FC denotes the fold change in protein abundance. The vertical dashed lines indicated log2(FC) thresholds, corresponding to |FC|≥1.5, respectively. The horizontal dashed line represented the P value threshold (P<0.05). Proteins with FC≥1.5 and P<0.05 were considered significantly upregulated in the GCNM group.
2.2 相互作用蛋白基因的GO和KEGG分析
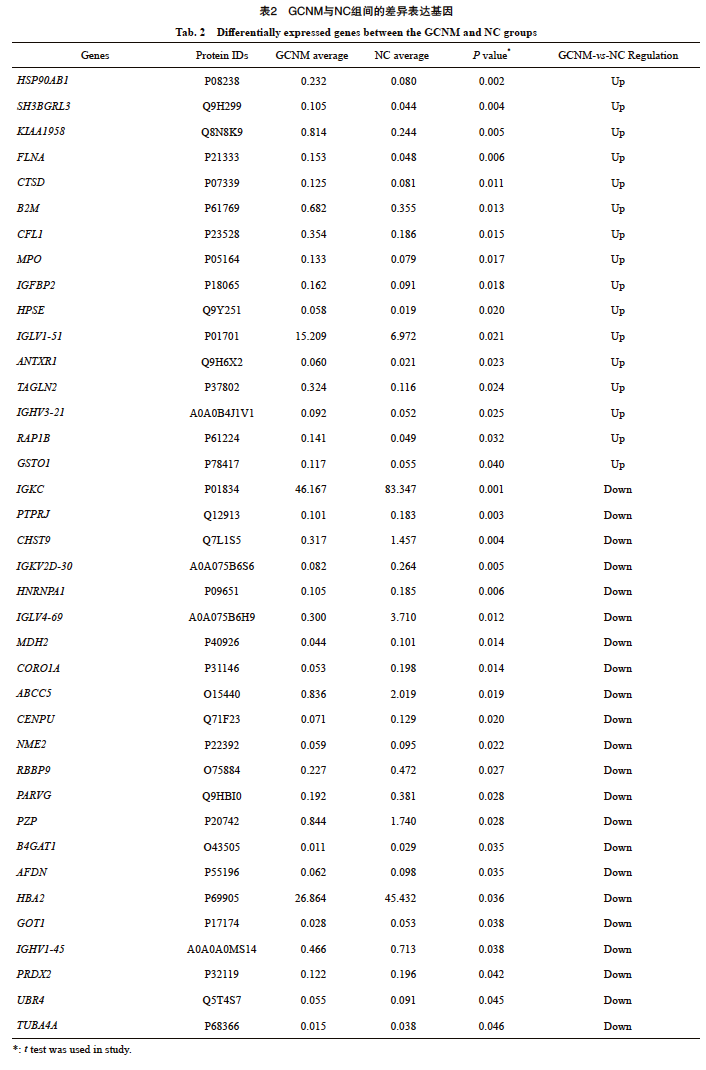
本研究基于STRING数据库对确定的16个上调基因构建了蛋白相互作用网络,用节点连接表示相互作用。在相互作用网络中发现了10个互作基因:TAGLN2、SH3BGRL3、CFL1、FLNA、HSP90AB1、CTSD、IGFBP2、GSTO1、B2M 和 MPO。对这10个基因进行GO和KEGG分析,并将结果进行可视化处理。KEGG信号转导通路分析显示了与肿瘤相关的信号转导通路,包括chemical carcinogenesis-receptor activation、NOD-like receptor signaling pathway、MAPK signaling pathway、estrogen signaling pathway、proteoglycans in cancer、 chemical carcinogenesis-DNA adducts等(图3A、B)。图3C展示了GO分析的30个富集术语,其中涵盖了与癌症发生、发展相关的条目:BP(如protein localization to cell surface、positive regulation of cell adhesion)、CC(如focal adhesion)和MF(如DNA polymerase binding)。富集结果表明,这10个基因可能与肿瘤进展有关。
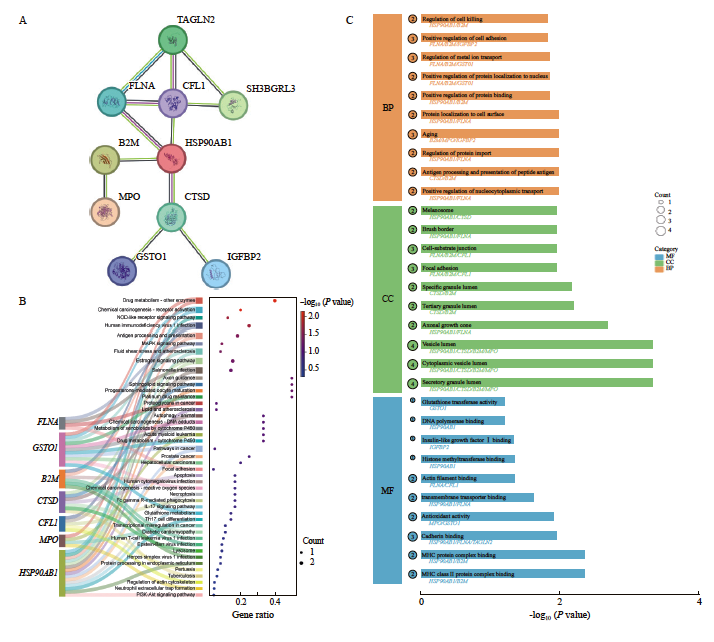
图3 表达上调的蛋白互作与GO和KEGG分析
Fig. 3 Protein-protein interactions and GO and KEGG analysis of upregulated proteins
A: Protein-protein interaction (PPI) network of differentially expressed proteins obtained through STRING analysis. B: KEGG pathway analysis of the top 10 interacting proteins. The dot size represented the number of genes associated with each enriched pathway. C: GO analysis of the top 10 interacting proteins. The dot size represented the number of genes associated with each enriched term. P<0.05 is considered statistically significant.
2.3 6种胃癌互作蛋白对应基因高表达且与患者预后不良有关
利用GEPIA 2数据库(包括408例胃癌和211例正常组织样本)验证了胃癌和正常组织中10个互作蛋白基因的差异表达。结果显示,胃癌组织高表达B2M、GSTO1、CTSD、CFL1、 TAGLN2、SH3BGRL3和HSP90AB1(图4A)。此外,Kaplan-Meier Plotter分析表明,B2M、IGFBP2、MPO、CFL1、TAGLN2、HSP90AB1、 SH3BGRL3、CTSD和FLNA的高表达与胃癌患者的不良预后相关(图4B)。其中6个枢纽基因(B2M、TAGLN2、CTSD、HSP90AB1、SH3BGRL3和CFL1)在胃癌中高表达且与患者预后不良相关。我们选择在胃癌中高表达且与不良预后相关的基因在后续临床组织样本中进一步验证它们作为胃癌潜在诊断标志物的潜力。
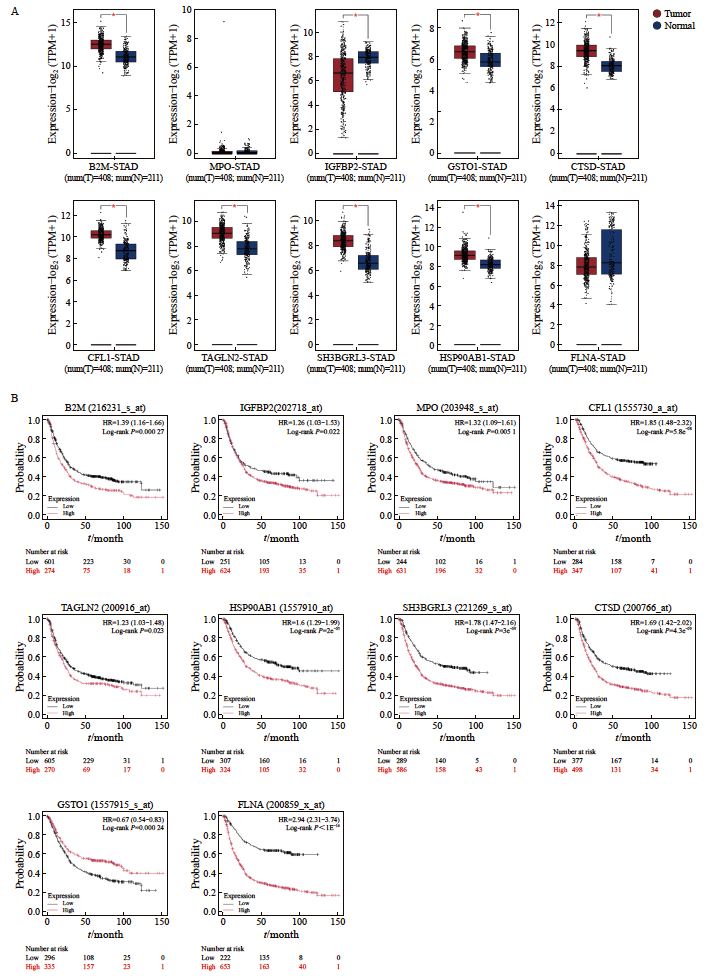
图4 胃癌组织中互作蛋白基因的表达差异及其与患者不良预后的关系
Fig. 4 Expression differences of interacting protein genes in gastric cancer tissues and their association with poor patient prognosis
A: Differential expression analysis of genes corresponding to interacting proteins between the gastric cancer group and the healthy group, based on TCGA and GTEx datasets using GEPIA 2. B: Correlation curves between interacting protein gene expression and overall survival in gastric cancer patients. HR>1 indicated a positive correlation, while HR<1 indicated a negative correlation. *: P<0.05, compared with each other.
2.4 qRT-PCR证实TAGLN2、CTSD、SH3BGRL3、CFL1和HSP90AB1在胃癌组织中高表达
统计分析结果表明,与癌旁组织相比,5种枢纽基因TAGLN2、CTSD、SH3BGRL3、CFL1和HSP90AB1在胃癌组织中呈高表达(图5)。空白对照组未检测到扩增产物。蛋白质组学、生物信息学和qRT-PCR分析最终确定TAGLN2、SH3BGRL3、CTSD、CFL1和HSP90AB1为诊断早期胃癌的潜在生物标志物。
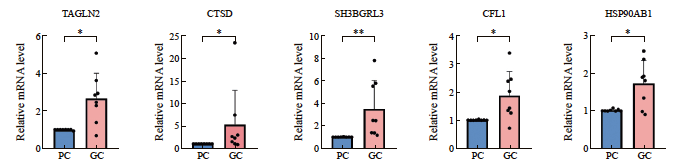
图5 胃癌与癌旁组织核心基因的qRT-PCR分析
Fig. 5 qRT-PCR analysis of core genes between the para-cancerous tissues and gastric tissues
PC: Para-cancerous tissues; GC: Gastric tissues. *: P<0.05, compared with each other; **: P<0.01, compared with each other.
3 讨 论
胃癌的发生是一个复杂的生物学过程,了解其分子机制并寻找更好的生物标志物对于胃癌的早期诊断和治疗至关重要。目前所用的胃癌早期诊断标志物并不理想,因此有必要寻找更灵敏、更特异的生物标志物。在本研究中,我们对GCNM患者和健康对照者的血清样本进行了蛋白质组学分析,以确定差异表达的蛋白质。qRT-PCR验证了这些基因在胃癌组织和癌旁组织中的表达,结果显示其中5种枢纽基因在胃癌中呈高表达。
基于质谱的蛋白质组学已广泛应用于生物医学研究。近年来,蛋白质组学研究从血清、组织和尿液样本中发现了胃癌的一些生物标志物[11]。在本研究中,我们对原发性胃癌患者和健康对照者的血清样本进行了定量分析,并进一步分析了与胃癌组中蛋白表达上调相对应的基因。GO和KEGG分析结果表明,枢纽基因的生物学过程和分子功能参与了胃癌的发生、发展。丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)和磷脂酰肌醇3-激酶(phosphatidylinositol 3-kinase,PI3K)信号转导通路已被证实参与胃癌的进展[12]。肌动蛋白细胞骨架的重塑促进胃癌细胞的迁移和侵袭[13],而钙调蛋白依赖性蛋白激酶2则通过MEK/细胞外信号调节激酶(extracellular signal-regulated kinase,ERK)信号转导通路激活胃癌细胞中的细胞周期蛋白依赖性激酶,促进其增殖[14]。GSTO1与活性氧(reactive oxygen species,ROS)相关,而ROS可作为诱导胃癌的化学致癌因子。然而,也有研究[15-16]表明,BDH2通过泛素化胃癌细胞中的Nrf2来促进ROS的积累,从而诱导细胞凋亡。代谢重编程诱导ROS积累,并依赖ROS恶性转化和氧化应激诱导凋亡效应[16],因此须进一步明确ROS在胃癌进展中的作用。
本研究发现,B2M、TAGLN2、CTSD、HSP90AB1、SH3BGRL3和CFL1是胃癌相关的枢纽基因,它们与肿瘤中免疫细胞浸润有关。既往研究[17]表明,枢纽基因在多种癌症中参与抑制抗肿瘤免疫。B2M编码β-2-微球蛋白,可诱导胶质母细胞瘤中的PI3K/蛋白激酶B(protein kinase B,AKT)信号转导通路激活,并通过上调转化生长因子-β1(transforming growth factor-β1,TGF-β1)促进M2型巨噬细胞极化。M2型巨噬细胞具有抗炎和促进肿瘤生长的作用。B2M的高表达也与肝细胞癌的进展有关[18]。B2M在转录水平上与程序性死亡受体配体1(programmed death-ligand 1,PD-L1)蛋白的表达水平呈正相关,并通过参与主要组织相容性复合体-Ⅰ(major histocompatibility complex-Ⅰ,MHC-Ⅰ)复合物的形成而促进淋巴细胞对肿瘤细胞的识别[19]。此外,肿瘤细胞表面表达的PD-L1与T淋巴细胞上的程序性死亡受体1(programmed death-1,PD-1)结合,抑制抗肿瘤免疫,PD-L1/PD-1遂成为免疫检查点抑制剂的治疗靶点。免疫治疗也能上调B2M的表达,表明基于PD-L1的免疫治疗效果良好[20]。主要在肿瘤细胞中表达的TAGLN2编码Transgelin-2,通过调节肌球蛋白细胞骨架、介导细胞黏附并增强肿瘤细胞迁移与侵袭[21-22],且与胃癌的微卫星不稳定性有关[23]。PI3K/AKT或MAPK/ERK信号转导通路诱导上皮-间质转化(epithelial-mesenchymal transition,EMT),通过NRP1/VEGFR2-MAPK信号诱导血管生成,并诱导胃癌细胞转移[24]。靶向TAGLN2可增强CD8+T淋巴细胞的细胞毒性,并介导T淋巴细胞与肿瘤细胞之间免疫突触的形成。肿瘤细胞分泌的Pro-cathepsin D可被间充质干细胞内化并转化为成熟的、具有催化活性的Cathepsin D,其能够抑制T淋巴细胞活化并促进M2巨噬细胞极化,参与补体C5处理和β-catenin活化,这些生物学过程均与肿瘤进展有关[25]。抑制CTSD可激活核因子κB(nuclear factor kappa-B,NF-κB)并使Bcl-xl失活,从而增加肿瘤对抗癌药物的敏感性[26]。HSP90AB1在多种癌症中高度表达,包括胃癌、肺腺癌和肝癌。具体而言,HSP90AB1可抑制泛素介导的LRP5降解,从而通过AKT和Wnt/β-catenin信号转导通路促进胃癌的EMT[27-28]。SH3BGRL3作为STAT3的上游基因,其表达可通过JAK-STAT3信号转导诱导胶质母细胞瘤,形成正反馈环路[29]。CFL1与TAGLN2一起通过细胞骨架重组介导EMT,从而导致胃癌的化疗耐药。低氧诱导因子-1α与其启动子上的低氧反应元件结合可激活CFL1的转录,CFL1可抑制泛素化,保护磷脂酶D1不被降解,最终诱导肝细胞的EMT。CFL1近年来已被认为是介导肿瘤化疗耐药的潜在靶点[30]。本研究筛选出的6种胃癌枢纽基因均参与肿瘤的发生、发展,与研究中的胃癌预后分析结果一致,具有作为胃癌诊断标志物的潜力。
综上所述,我们基于血清蛋白质组学发现并验证了5种潜在的早期胃癌诊断生物标志物:TAGLN2、CTSD、HSP90AB1、SH3BGRL3和CFL1。这些基因在早期胃癌患者的血清和组织中均高度表达。本研究为了解胃癌进展的分子机制提供了新的视角,未来的研究应侧重于阐明这些基因影响免疫浸润和肿瘤进展的确切分子机制,并探索它们在临床应用中的效能,以改善胃癌的早期诊断和治疗。相比于多中心大型研究,本研究样本基数小,且本研究未完全控制年龄、性别、饮食习惯等潜在混杂因素,这些因素可能影响血清中标志物的表达水平,导致结果的偏倚。此外,本研究未对标志物的诊断效能如灵敏度、特异度进行全面的评估,也缺少与传统标志物联合使用的对比研究,可能忽视了标志物在实际临床应用中的局限性。我们将在后续研究中完善临床资料、扩大标本量,通过进一步的队列研究验证其相较或联合传统血清肿瘤标志物在胃癌诊断中的应用价值。
致谢:
感谢STRING数据库、GEPIA 2数据库和Kaplan-Meier Plotter提供宝贵的数据集用于统计分析。感谢陈明杰(上海纽科生物科技有限公司)提供数据分析和可视化支持。同时,我们也感谢兰州大学第二医院临床医学院萃英生物医学研究中心提供的技术支持。
第一作者:
杜可为,兰州大学第二临床医学院在读硕士研究生。
通信作者:
尤崇革,教授,主任检验技师,博士研究生导师。
作者贡献声明:
杜可为进行了实验分析,文献调研与整理,绘制图表并起草了论文;张尚弟设计研究思路,提出研究方向与论文框架,设计了研究方案并修订了论文;胡文飞、高山、甘建新收集临床标本并提供技术指导;尤崇革监督实验过程并完成初稿、修订稿的修订和审阅,负责投稿事宜。所有作者都阅读并通过了最终稿件。
[参考文献]
[1] THRIFT A P, WENKER T N, EL-SERAG H B. Global burden of gastric cancer: epidemiological trends, risk factors, screening and prevention[J]. Nat Rev Clin Oncol, 2023, 20(5): 338-349.
[2] WONG M C S, HUANG J J, CHAN P S F, et al. Global incidence and mortality of gastric cancer, 1980-2018[J]. JAMA Netw Open, 2021, 4(7): e2118457.
[3] JELSKI W, MROCZKO B. Molecular and circulating biomarkers of gastric cancer[J]. Int J Mol Sci, 2022, 23(14): 7588.
[4] SHEN N, ZHU S S, ZHANG Z Y, et al. High expression of COL10A1 is an independent predictive poor prognostic biomarker and associated with immune infiltration in advanced gastric cancer microenvironment[J]. J Oncol, 2022, 2022: 1463316.
[5] FENG L R, LI G X, LI D B, et al. Cuproptosis-related gene SERPINE1 is a prognostic biomarker and correlated with immune infiltrates in gastric cancer[J]. J Cancer Res Clin Oncol, 2023, 149(12): 10851-10865.
[6] 中华人民共和国国家卫生健康委员会医政医管局, 季加孚.胃癌诊疗指南(2022年版)[J]. 中华消化外科杂志, 2022, 21(9): 1137-1164.
Medical Administration Bureau of the National Health Commission of the People's Republic of China, JI J F. Standardization for diagnosis and treatment of gastric cancer(2022 edition)[J]. Chin J Dig Surg, 2022, 21(9): 1137-1164.
[7] CHEN T, MA J, LIU Y, et al. iProX in 2021: connecting proteomics data sharing with big data[J]. Nucleic Acids Res, 2022, 50(D1): D1522-D1527.
[8] SZKLARCZYK D, KIRSCH R, KOUTROULI M, et al. The STRING database in 2023: protein-protein association networks and functional enrichment analyses for any sequenced genome of interest[J]. Nucleic Acids Res, 2023, 51(D1): D638-D646.
[9] GYŐRFFY B. Integrated analysis of public datasets for the discovery and validation of survival-associated genes in solid tumors[J]. Innovation (Camb), 2024, 5(3): 100625.
[10] TANG D D, CHEN M J, HUANG X H, et al. SRplot: a free online platform for data visualization and graphing[J]. PLoS One, 2023, 18(11): e0294236.
[11] FENG T, JIE M W, DENG K, et al. Targeted plasma proteomic analysis uncovers a high-performance biomarker panel for early diagnosis of gastric cancer[J]. Clin Chim Acta, 2024, 558: 119675.
[12] MORGOS D T, STEFANI C, MIRICESCU D, et al. Targeting PI3K/AKT/mTOR and MAPK signaling pathways in gastric cancer[J]. Int J Mol Sci, 2024, 25(3): 1848.
[13] WANG Y L, TU Z Y, ZHAO W, et al. PLCB1 enhances cell migration and invasion in gastric cancer via regulating actin cytoskeletal remodeling and epithelial-mesenchymal transition[J]. Biochem Genet, 2023, 61(6): 2618-2632.
[14] NAJAR M A, ARAVIND A, DAGAMAJALU S, et al. Hyperactivation of MEK/ERK pathway by Ca2+/calmodulindependent protein kinase kinase 2 promotes cellular proliferation by activating cyclin-dependent kinases and minichromosome maintenance protein in gastric cancer cells[J]. Mol Carcinog, 2021, 60(11): 769-783.
[15] WANG Q, XU C, FAN Q, et al. Positive feedback between ROS and cis-axis of PIASxα/p38α-SUMOylation/MK2 facilitates gastric cancer metastasis[J]. Cell Death Dis, 2021, 12(11): 986.
[16] WANG Y W, QI H, LIU Y, et al. The double-edged roles of ROS in cancer prevention and therapy[J]. Theranostics, 2021, 11(10): 4839-4857.
[17] LI D Q, ZHANG Q, LI L, et al. β2-microglobulin maintains glioblastoma stem cells and induces M2-like polarization of tumor-associated macrophages[J]. Cancer Res, 2022, 82(18): 3321-3334.
[18] LIN Q M, JIANG Z W, MO D, et al. Beta2-microglobulin as predictive biomarkers in the prognosis of hepatocellular carcinoma and development of a new nomogram[J]. J Hepatocell Carcinoma, 2023, 10: 1813-1825.
[19] WANG H B, LIU B R, WEI J. Beta2-microglobulin(B2M) in cancer immunotherapies: biological function, resistance and remedy[J]. Cancer Lett, 2021, 517: 96-104.
[20] REIS B, ATTIG J, DZIADEK S, et al. Tumor beta2- microglobulin and HLA-a expression is increased by immunotherapy and can predict response to CIT in association with other biomarkers[J]. Front Immunol, 2024, 15: 1285049.
[21] YIN L M, ULLOA L, YANG Y Q. Transgelin-2: biochemical and clinical implications in cancer and asthma[J]. Trends Biochem Sci, 2019, 44(10): 885-896.
[22] JI C B, ZHAO J J, CHEN H, et al. Single-cell RNA sequencing reveals the lineage of malignant epithelial cells and upregulation of TAGLN2 promotes peritoneal metastasis in gastric cancer[J]. Clin Transl Oncol, 2023, 25(12): 3405-3419.
[23] PAN T, WANG S B, WANG Z Y. An integrated analysis identified TAGLN2 as an oncogene indicator related to prognosis and immunity in pan-cancer[J]. J Cancer, 2023, 14(10): 1809-1836.
[24] LEE S G, WOO S M, SEO S U, et al. Cathepsin D promotes polarization of tumor-associated macrophages and metastasis through TGFBI-CCL20 signaling[J]. Exp Mol Med, 2024, 56(2): 383-394.
[25] SEO S U, WOO S M, IM S S, et al. Cathepsin D as a potential therapeutic target to enhance anticancer drug-induced apoptosis via RNF183-mediated destabilization of Bcl-xL in cancer cells[J]. Cell Death Dis, 2022, 13(2): 115.
[26] WANG H N, DENG G X, AI M L, et al. Hsp90ab1 stabilizes LRP5 to promote epithelial-mesenchymal transition via activating of AKT and Wnt/β-catenin signaling pathways in gastric cancer progression[J]. Oncogene, 2019, 38(9): 1489-1507.
[27] JIA L Q, GE X L, DU C, et al. EEF1A2 interacts with HSP90AB1 to promote lung adenocarcinoma metastasis via enhancing TGF-β/SMAD signalling[J]. Br J Cancer, 2021, 124(7): 1301-1311.
[28] LIN X, LIU Y H, ZHANG H Q, et al. DSCC1 interacts with HSP90AB1 and promotes the progression of lung adenocarcinoma via regulating ER stress[J]. Cancer Cell Int, 2023, 23(1): 208.
[29] NIE Z, CHENG D T, PAN C L, et al. SH3BGRL3, transcribed by STAT3, facilitates glioblastoma tumorigenesis by activating STAT3 signaling[J]. Biochem Biophys Res Commun, 2021, 556: 114-120.
[30] SOUSA-SQUIAVINATO A C M, MORGADO-D.AZ J A. A glimpse into cofilin-1 role in cancer therapy: a potential target to improve clinical outcomes? [J]. Biochim Biophys Acta Rev Cancer, 2024, 1879(2): 189087.
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)