首页 > 医疗资讯/ 正文
蛋白质是生命活动主要承担者,通过与其他蛋白质或肽的相互作用实现其生物学功能。近年来,深度学习的迅速发展彻底改变了各个领域的预测工作,蛋白质语言模型(pLMs)不仅被用于设计功能类似天然蛋白质的合成蛋白质序列,还能预测蛋白质结构,并在预测抗体进化等特定场景中发挥作用。
现有pLMs在表征蛋白质序列时表现较好,但在解析蛋白质相互作用的复杂模式时存在局限,尤其对于肽段-主要组织相容性复合体(pMHC)相互作用的预测,传统工具需特定等位基因的训练数据,难以应对跨类型、跨物种的零样本预测任务。
为解决上述难题,美国匹兹堡大学研究团队开发了一种交互语言模型(iLM)——SWING,其通过挖掘氨基酸特性差异构建交互词汇,为解决蛋白质相互作用预测难题提供了新方案。SWING能精准预测I类和II类 pMHC相互作用,可跨类型(I类模型预测II类)、跨物种(人类模型预测小鼠)进行预测。在变异效应解析方面,SWING仅凭序列信息即可准确识别错义变异对特定蛋白质相互作用的破坏机制。总之,凭借独特的滑动窗口架构,SWING实现了蛋白质相互作用语言的零样本学习,为蛋白质互作研究提供了无需领域特化训练的通用型解决方案。

SWING的设计突破了传统pLMs的局限,其核心在于直接编码蛋白质间的残基配对信息,而非对单个蛋白质的嵌入特征进行后处理。具体而言,模型包括以下步骤:
滑动窗口机制
选取n长度肽段作为滑动窗口,从靶序列首位开始逐位滑动,计算每个位点氨基酸对在生化指标(如极性、疏水性)上的绝对差异并量化编码。该机制直接捕获互作相关的局部生化信息。
交互词汇库构建
将编码后的序列拆分为重叠k-mer片段,每个k-mer视为"词汇",将整个交互过程视为由这些词汇组成的"文档"。这种设计使模型能全面采样具有生化意义的残基对组合。
嵌入生成架构
利用Doc2Vec模型将整个相互作用转化为交互嵌入向量,作为下游预测器(如XGBoost)的特征输入,从而完成具体预测任务(如pMHC结合预测、变异影响预测等)。这种架构无需依赖单个蛋白质的预训练嵌入,能直接捕捉相互作用的关键信息,且对序列长度不敏感,适用于多种交互场景。
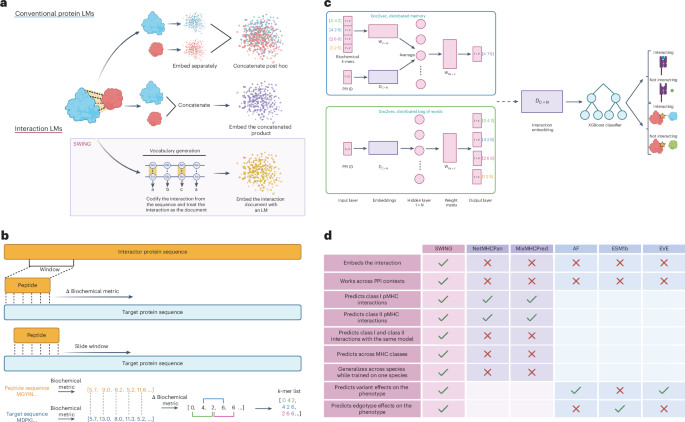
图1. SWING概述
MHC分子(MHC-I和MHC-II)与肽的相互作用(pMHC)是免疫应答的关键环节,但因MHC等位基因多样、可能的组合数量庞大等难以通过实验进行表征。SWING在pMHC相互作用预测任务中展现出卓越性能。交叉验证显示,在人类pMHC-I中,SWING的AUC达0.72,对未知等位基因(如HLA-A02:02)预测稳定,AUC保持在0.63-0.84;针对pMHC-II,模型AUC提升至0.90,对未知等位基因(如DRB1_0102)预测时AUC达0.93-0.95。
研究团队还通过三重验证实验分析了影响SWING性能的关键因素。结果显示,该模型不受特定生化指标和MHC序列长度的影响。特别地,当肽长度小于9个氨基酸(9-AA )时,因关键结合信息丢失,模型性能急剧下降,这与已知的MHC结合核心区域生物学特性一致。
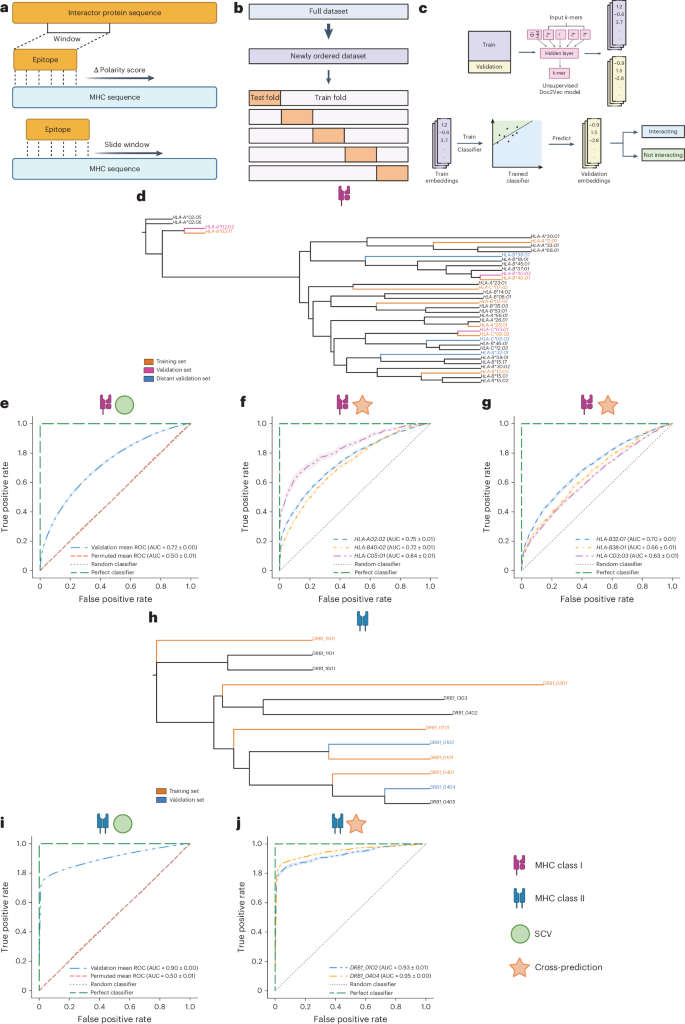
图2. SWING预测pMHC
少样本和零样本学习通过重用“数据丰富” 场景中的已有知识,来解决 “数据稀疏” 场景中相关的问题, SWING的嵌入包含相互作用特异性信息可能遵循这一原理,为此研究团队验证其跨类别与跨物种预测能力。 结果显示,I类SWING模型能准确预测II类pMHC相互作用,AUC为0.74-0.77,性能显著优于NetMHCpan等基准方法。此外,研究团队还在 I、II类数据上训练了SWING联合预测模型,突破II类pMHC数据稀缺限制,能有效预测两类pMHC相互作用,性能稳定且未受单一类别数据影响。
基于人类MHC数据训练的SWING模型,可精准预测鼠源II类MHC与自身免疫病风险相关等位基因的相互作用。对于H-2-IEk(与系统性红斑狼疮相关),SWING预测性能全面超越MixMHC2pred与NetMHCIIpan,且对不同长度肽段召回率较高,无明显长度偏差。对于H-2-IAg7(与1型糖尿病相关),SWING的零样本预测召回率最高,而其他工具表现欠佳。
在全新数据和人类罕见疾病等位基因的预测中,SWING不仅性能与现有工具相当或更优(AUC为0.81-0.84),还能覆盖所有已知肽段长度范围,尤其在新等位基因和非标准肽段的预测上弥补了现有方法的不足,充分体现了其独特的实用性和广泛适用性。
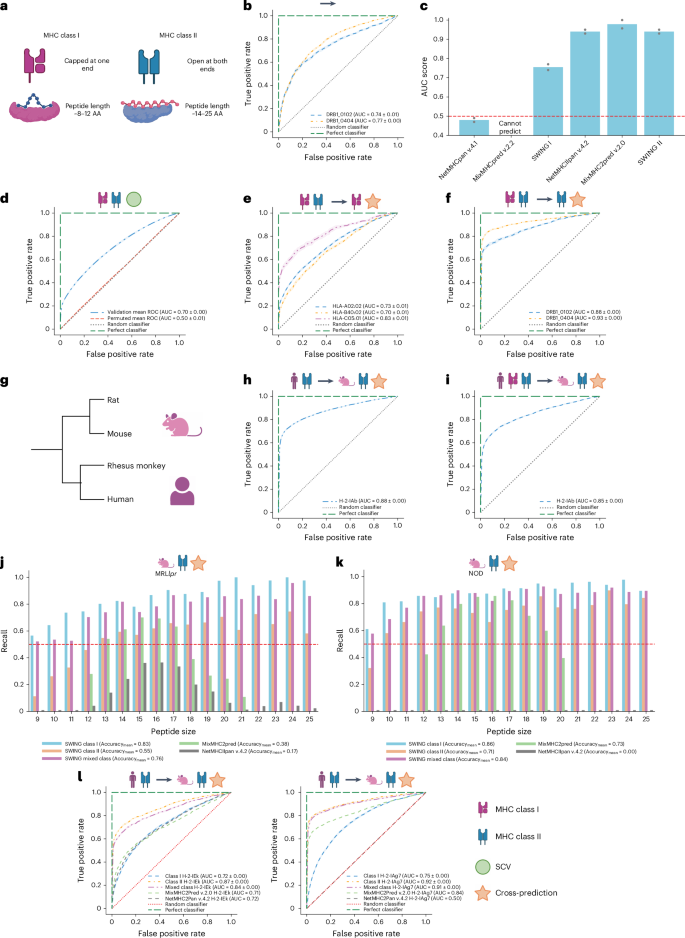
图3. 零样本SWING性能
变异效应预测(VEP)通常在生物体层面进行,目前多数工具聚焦于致病性判断,难以评估其对特定蛋白质相互作用的破坏。SWING填补了这一空白,成功预测了错义变异对蛋白质相互作用的破坏效应。SWING在预测孟德尔疾病相关变异、人群常见和罕见变异对相互作用的影响时,表现出高准确性,AUC分别为0.87和0.80,显著超越AlphaMissense等预测工具;模型的预测性能与生化指标、分类器类型无关。此外,SWING预测结果与次等位基因频率呈负相关。
研究团队重点介绍了SWING预测的两个破坏性变异,一个是东亚人群常见变异(PRKCH基因rs2230500变异),影响PRKCH与BANP的相互作用,另一个(HPX基因rs12117变异)可能破坏HPX与GLUD1的相互作用。上述结果表明SWING在识别变异引起的相互作用破坏方面具有重要潜力,超越了传统的VEP方法。
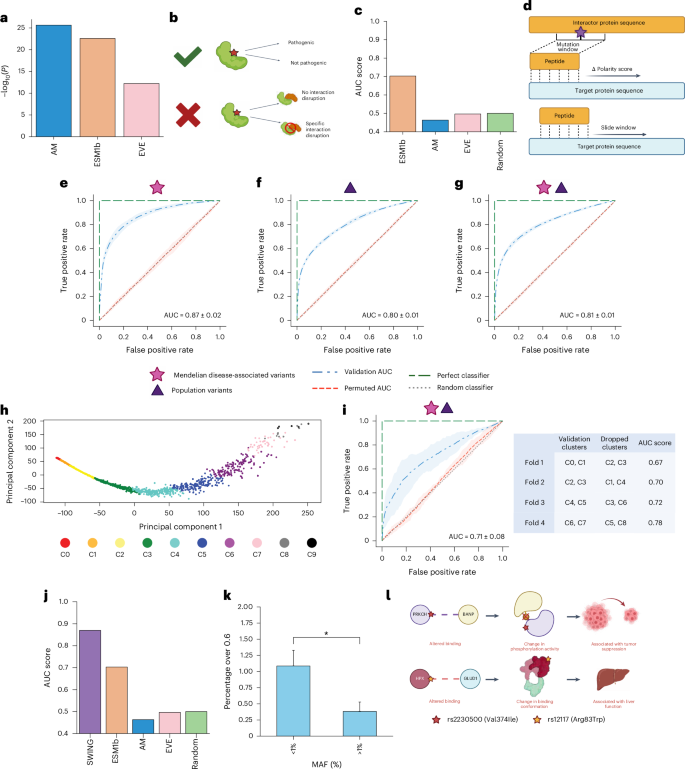
图4. SWING预测相互作用破坏
最后,研究团队将SWING与其他多种模型进行了性能比较。在多项任务中,SWING 均优于现有方法。在跨类型、跨物种场景中,SWING是唯一能够有效预测pMHC相互作用的模型。在错义变异效应预测中,SWING的AUC为0.81,显著高于CNN-iLM、BERT-iLM等模型。此外,无论生化指标如何选择,SWING性能均保持稳定,表明其捕捉的是相互作用的本质特征,而非特定指标的偏差。
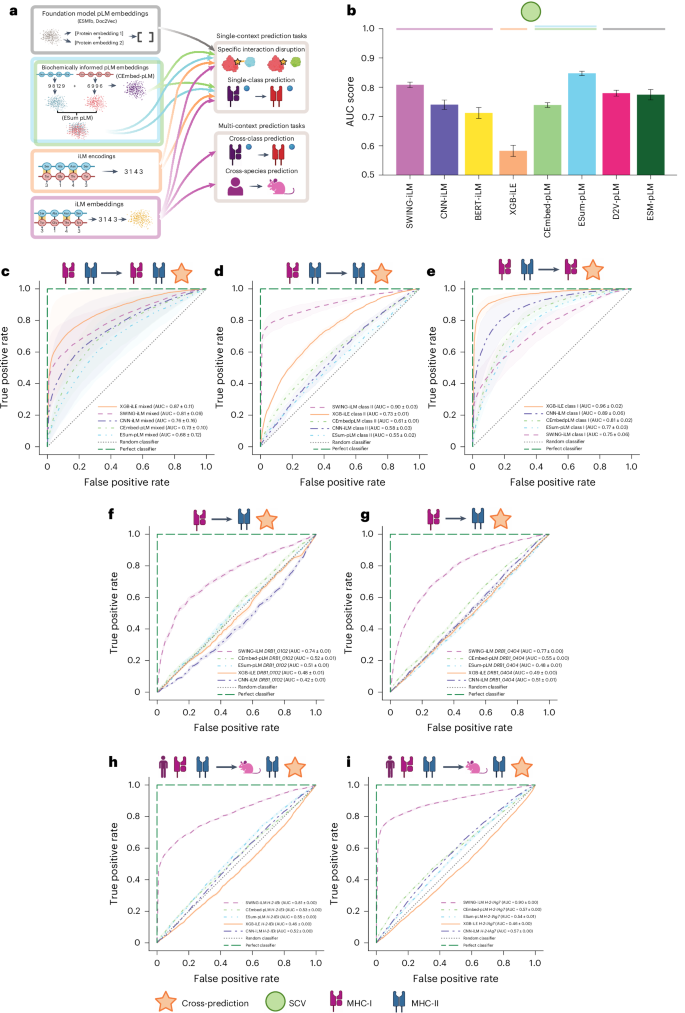
图5. 多种交互建模方法的比较
综上所述,SWING模型是一种不受序列长度限制的相互作用语言模型,能捕捉蛋白质-蛋白质或蛋白质-肽的相互作用。该模型通过极性/疏水性差异编码残基接触规则,规避结构信息依赖;实现I/II类MHC零样本交叉预测,助力罕见等位基因研究。SWING 的零样本泛化能力和对序列长度的灵活性,使其在数据稀缺场景中更具优势,有望成为蛋白质交互研究的重要基础工具。
参考文献:
Siwek, J.C., Omelchenko, A.A., Chhibbar, P. et al. Sliding Window Interaction Grammar (SWING): a generalized interaction language model for peptide and protein interactions. Nat Methods (2025). https://doi.org/10.1038/s41592-025-02723-1
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)