首页 > 医疗资讯/ 正文
今年8月,谷歌旗下的英国DeepMind公司借助AlphaFold2模型,公布了大约 2.2 亿种蛋白质的预测结构,它几乎涵盖了 DNA 数据库中已知生物体的所有蛋白质。AlphaFold是曾开发出AlphaGo的DeepMind公司研发的蛋白质结构预测深度学习模型,见:谷歌AlphaFold破译“上帝密码”,解答困扰人类50年难题。2021年7月,DeepMind表示,AlphaFold2与AlphaFold完全不同,“这是一个基于神经网络的新模型,其预测的蛋白质结构能达到原子水平的准确度。”这一巨大进步被Nature和Science选为“2021年度十大科学突破”。详细见:“AlphaFold像地震一样,改变了游戏规则”,如今它开始在基础研究、药物开发上全面发力
而现在,另一家科技巨头Meta(前身为Facebook)正借助人工智能ESMFold(“宏基因组”数据库) 填补我们蛋白质宇宙的“暗物质”。
科技公司Meta开发的ESMFold成功预测了超过6亿个蛋白质三维结构,包含大量前所未见的结构,并且预测速度最高可达AlphaFold的60倍。这些数据组成了全球首个大规模的宏基因组蛋白质结构图谱,推动人类对蛋白质结构的理解进入全新时代。该研究目前上线于预印本平台biorxiv。

AlphaFold2主要利用MSA(多序列比对)信息,把蛋白质的结构和生物信息整合到了深度学习算法中,所以当面对比如孤儿序列(找不到其他与其相像的序列)这样的蛋白时,该模型就无法准确地生效了。
在土壤中、海水里甚至是我们体内,各种微生物无处不在。除了那些已经被科学家分类、注释的生命,未知的微生物中还含有更多无人知晓的蛋白质结构。宏基因组研究正是对这些环境样本进行测序,从而寻找其中的全新蛋白质。在最新研究中,ESMFold的应用对象就是宏基因组DNA数据库。
虽然ESMFold和AlphaFold都是预测蛋白质结构的工具,但两者的策略完全不同。AlphaFold的预测是基于多序列比对,需要更多额外信息;而ESMFold仅仅基于氨基酸序列,就能进行结构预测。
令人意外的是,ESMFold使用的,是一款看似毫不相干的大型语言模型。语言模型通过部分字母、单词来预测文本,而在研究团队看来,语言模型与蛋白质预测的逻辑有着相通之处。
一段文字可以拆解为一个个字母序列,而蛋白质结构同样由20个“字母”,也就是氨基酸的序列构成。另一方面,文字的理解需要结合上下文语境,而在氨基酸序列折叠成三维结构的过程中,当两个特定的氨基酸配对出现,意味着在三维结构中这两个位点就如同两块能拼在一起的拼图,很可能存在相互作用。
因此,研究团队需要做的就是通过两个步骤训练语言模型,使其学会根据氨基酸序列解读蛋白质结构。
团队负责人Alexander Rives表示,这些训练让ESMFold对包含蛋白质形状信息的蛋白质序列有了直观了解。而且,与“阿尔法折叠”一样,这一网络能将这些了解到的信息与已知蛋白质结构和序列之间关系的信息结合,生成预测结构。ESMFold虽然不像“阿尔法折叠”那么准确,但在预测结构方面的速度要快60倍,这意味着他们可将结构预测扩展到更大的数据库。Alexanderander Rives称:“这些结构是我们所知最少的,我认为它们提供了深入了解生物学的潜力。该模型的准确性水平可以与AlphaFold2‘竞争’”。
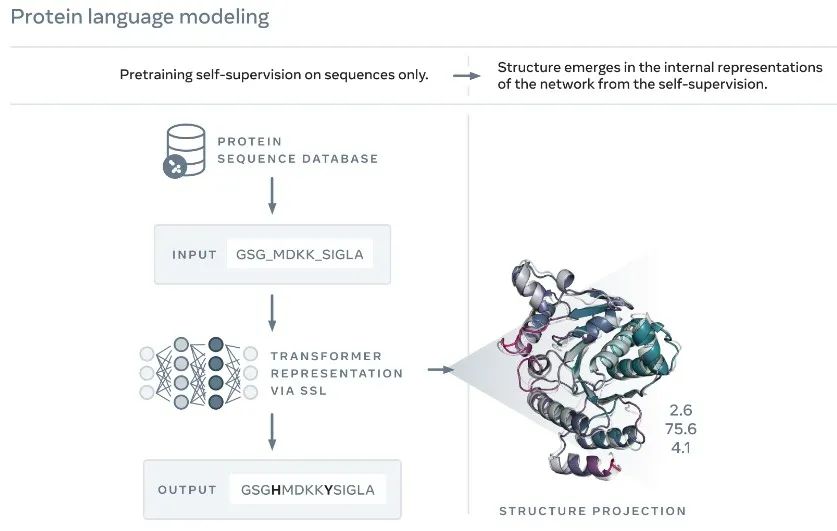 ▲研究团队通过训练语言模型,实现对蛋白质结构的预测(图片来源:参考资料[2])
▲研究团队通过训练语言模型,实现对蛋白质结构的预测(图片来源:参考资料[2])
他们首先将大量已知蛋白质的氨基酸序列输入模型,并特意留出一些空白。而语言模型就像是处理文本信息一样,基于直觉形成对氨基酸序列的理解,并自动填补空白、得到完整的序列。
在此基础上,研究团队基于蛋白质数据库中的大量结构,通过监督学习训练模型预测蛋白质三维结构的能力。值得一提的是,这个过程利用了AlphaFold的预测,以提升模型的预测能力。最终,Meta AI团队创建了迄今最大的蛋白质语言模型,并且能够以原子分辨率预测蛋白质结构。
由于ESMFold是直接基于氨基酸序列进行预测,相比于AlphaFold,预测流程得到了简化。其直接体现就是速度:其预测蛋白质结构的速度最高可达AlphaFold的60倍。“这意味着,我们可以将对蛋白质结构的预测扩展至更大的数据库中。”论文作者,Meta AI研究团队的领导者Alexander Rives博士说。但与此同时,ESMFold预测的准确率不及AlphaFold。
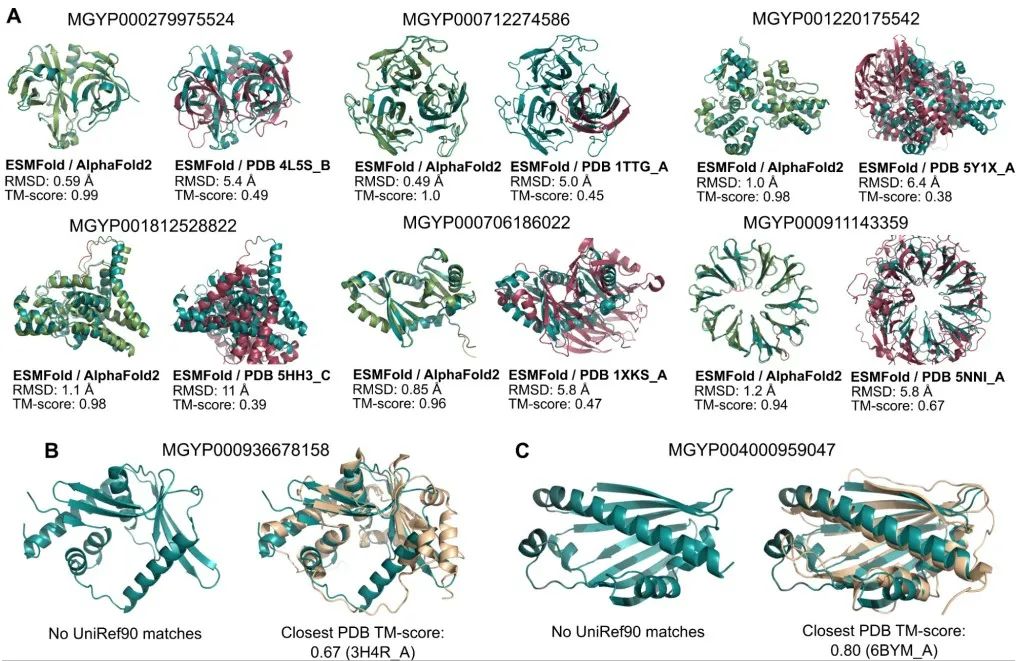 ESMFold预测出的部分蛋白质结构(图片来源:参考资料[1])
ESMFold预测出的部分蛋白质结构(图片来源:参考资料[1])
利用ESMFold,研究团队对来自环境土壤、海水、人的肠道与皮肤以及其他微生物栖息场所的样本进行了宏基因组DNA测序,仅用两周时间就预测出超过6.17亿个蛋白质结构。其中至少有2.25亿属于高精度预测:整体蛋白质形态正确,部分情况下可识别原子层面的细节。
在这些能进行进一步分析的高精度预测结构中,有76.8%都与数据库中已有的蛋白质结构存在显著差异,而12.6%更是与实验确定的结构完全不匹配。这些结果意味着,宏基因组中储存着大量前所未见的蛋白质结构。
宏基因组数据库“应该涵盖了此前未知的蛋白质宇宙中的一大部分,”未参与该研究的首尔大学计算生物学家Martin Steinegger教授说,“这项研究为探索更多黑暗的角落提供了巨大机遇。”
数十亿年前,生命演化出的蛋白质语言构成了复杂、动态的分子机器。而学会解读蛋白质语言,是理解自然世界的重要一步。
就如同显微镜的诞生,AI的参与也让科学家能在前所未有的小尺度上理解生命过程。而基于语言模型诞生的ESMFold,让人们拥有了更简洁、简单且廉价的工具,得以深入解读复杂的蛋白质语言,探索这些未知蛋白质的功能,甚至找到新的物种。“对于这些神秘的蛋白质我们所知甚少,我想这项发现为深入理解生物学提供了潜力。”Rives博士说。
不过,也有科学家对其预测的精准度表示怀疑。哈佛大学进化生物学家Sergey Ovchinnikov认为其中有些预测结果可能缺乏明确的结构,另一些可能属于被误判的非编码DNA。“似乎仍有一半以上的蛋白质空间我们一无所知。”他说。德国慕尼黑工业大学的计算生物学家Burkhard Rost也质疑:ESMFold模型在预测宏基因组数据库中的蛋白质时,是否真的比AlphaFold2精度更具优势。
对此,澎湃科技连线了复旦大学复杂体系多尺度研究院院长马剑鹏。马剑鹏表示,虽然ESMFold的预测准确性尚未达到AlphaFold,但在预测结构方面它比 AlphaFold 快约 60 倍,这意味着其可以用于构建更大的蛋白质预测结构数据库。
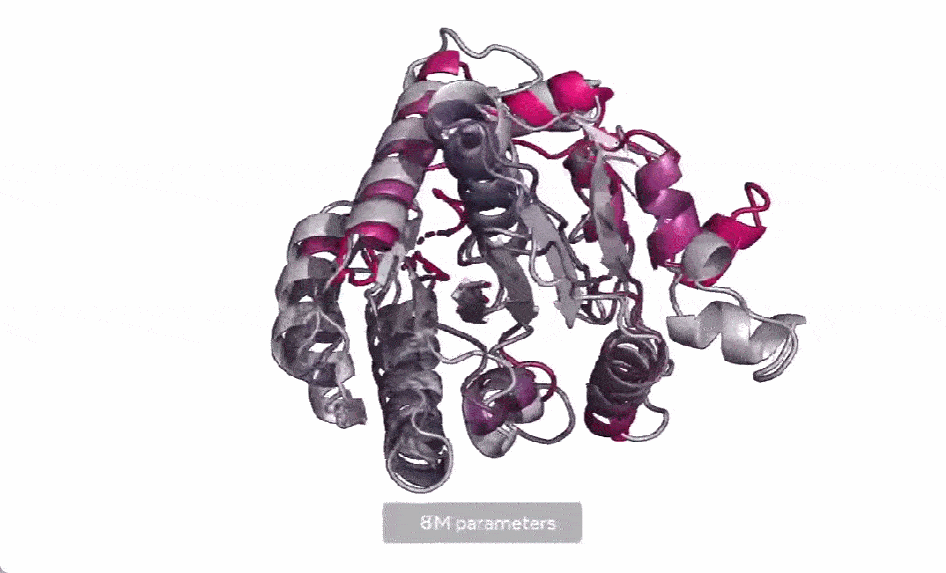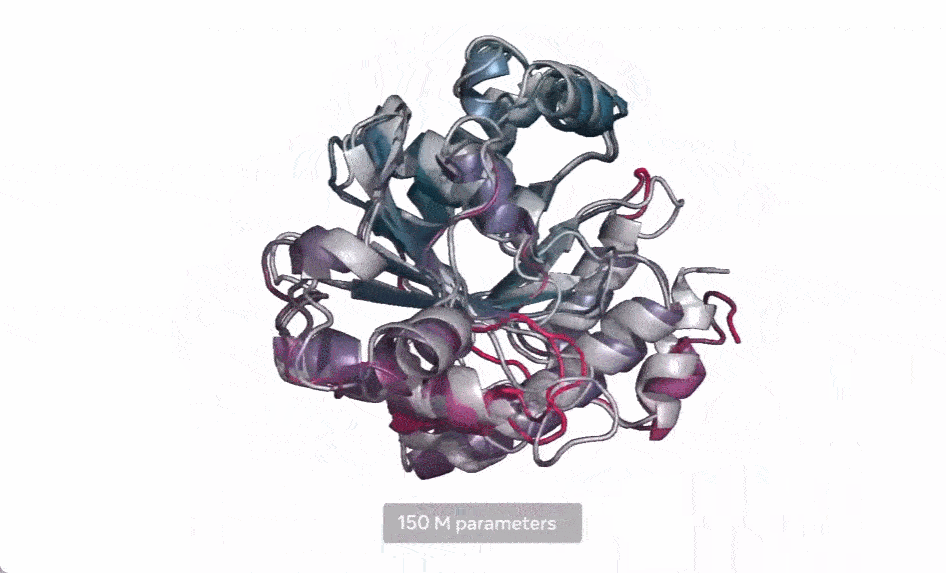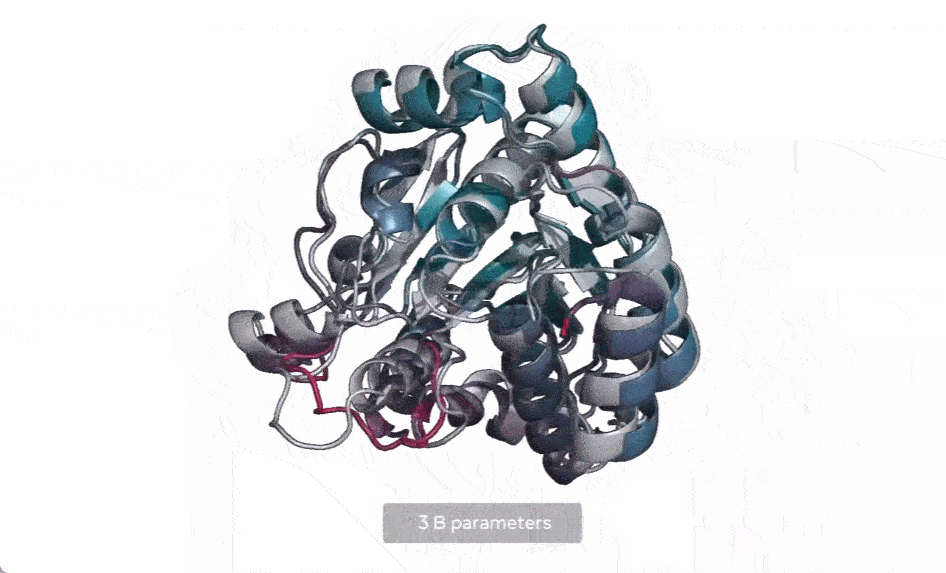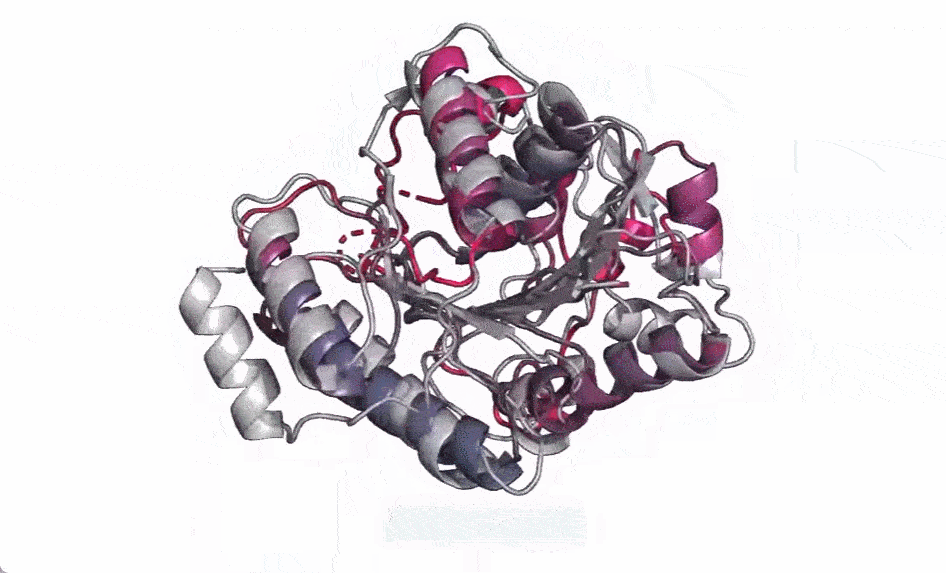
马剑鹏认为,该模型的贡献还在于向世界展示了AI算法的强大,连预测蛋白质结构这种困扰了人类几十年的难题都可以借助AI解决,说明一个属于AI算法的时代已经离我们不远了。
当然,ESMFold助推新药研发的话,这是一个需要时间的过程。本质上来讲AlphaFold2和ESMFold模型的底层逻辑是一致的,他们主要预测蛋白质的静态结构,下一步还需要研究蛋白质的动态结构,这个研究领域叫分子动力学模拟。所以该模型也会帮助我们加快了分子动力学模拟领域的研究速度。当这两个领域的研究都完成后,下一步才是药物设计。总之,AI赋能新药设计这个领域从长远看确实具有巨大的潜力,但是还需要一步一个脚印前进,还需要时间。
参考资料:
Lin, Z. et al. Preprint at BioRxiv, https://www.biorxiv.org/content/10.1101/2022.07.20.500902v2 (2022).https://www.thepaper.cn/newsDetail_forward_20562107
https://mp.weixin.qq.com/s/Ac9qv0DbzWNA3PeXDF-f-g
ESM Metagenomic Atlas: The first view of the ‘dark matter’ of the protein universe. Retrieved Nov 1st, 2022 from https://ai.facebook.com/blog/protein-folding-esmfold-metagenomics/
AlphaFold’s new rival? Meta AI predicts shape of 600 million proteins. Retrieved Nov 1st, 2022 from https://www.nature.com/articles/d41586-022-03539-1
https://www.medsci.cn/article/show_article.do?id=8ae7e342705f
https://baijiahao.baidu.com/s?id=1748369100573065865&wfr=spider&for=pc
https://ai.facebook.com/blog/protein-folding-esmfold-metagenomics/
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)