首页 > 医疗资讯/ 正文
导读
英国生物样本库(UKB)是全球最重要的健康研究资源之一,已用于多模态脑成像、蛋白质组学、代谢组学等大量数据收集和研究,显著增加了其数据集的深度。在最初的版本中,UKB所有样本都进行了基因分型,并推算出约9600万个单核苷酸多态性(SNP)。SNP基因分型可以准确表征相对常见的变异,但不适合罕见遗传变异和基因组的复杂区域。同时,UKB样本还进行了全外显子组测序(WES),但忽略了几乎所有的非编码变异,并且在检测SV方面受到限制。此外,有研究表明罕见的非编码变异对人类疾病和其他复杂特征有影响,但相关研究仍相对不足。
全基因组测序(WGS)通过提供人类基因组的全面图谱,突破了传统基因分型技术的局限,为遗传变异识别开辟了新途径,可以扩展对罕见非编码变异在健康和疾病中的作用的理解。
近日,UKB WGS联盟报告了490640名UKB参与者的WGS最新结果,加深了我们对遗传学如何与疾病生物学相关联的理解,并进一步提高了该开放资源对人类生物学和健康研究的价值。该研究发现了约15亿个变异,包括SNP、插入缺失(Indel)和结构变异(SV)。这些变异中的许多与各种疾病特征和性状有关,并可能有助于更深入地理解疾病机制,包括通过非编码机制影响疾病风险。同时,该研究通过分析不同族裔群体的基因组关联性及跨族裔关联,结合丰富的表型数据,揭示了新的遗传学和临床见解。虽然疾病特征相关的大多数关联主要出现在欧洲裔人群中(其中93.5%为非芬兰欧洲裔,其余31,785例来自其他大陆族群),但非洲和亚洲裔人群也显示出显著或全新的信号。

文章发表在Nature
该数据集作为UKB研究可获取的大型WGS资源库,将推动人类基因组认知的突破性进展,助力开发疗效更优、安全性更高的诊断与治疗方案,并为精准医疗策略提供创新路径,有望显著改善全球公共卫生水平。
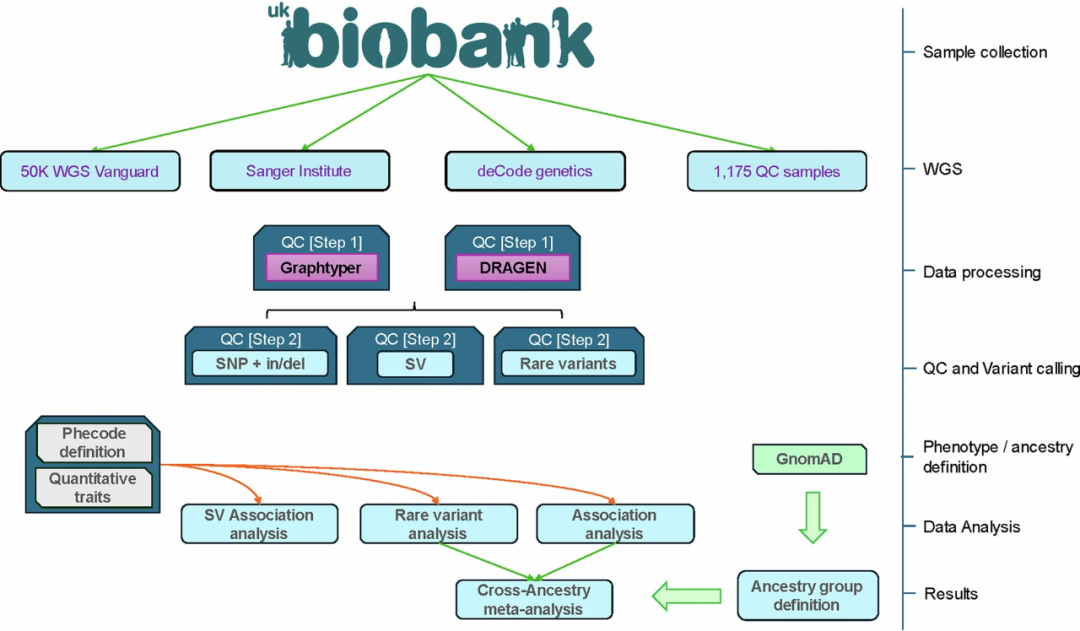
图1.UKB研究框架
主要研究内容
作为UKB最新一轮数据采集的重要组成部分,研究团队使用Illumina NovaSeq 6000测序仪对490,640名参与者进行了WGS,平均总测序深度达32.5x(单个样本最低23.5x)。为确保数据质量,其中1,175份样本还进行了两次测序验证。研究团队在UKB WGS数据集中定义了5个具有不同祖先的队列,包括非洲裔(AFR)、犹太裔(ASJ)、东亚裔(EAS)、非芬兰欧洲裔(NFE)和南亚裔(SAS)。
WGS在UKB参与者中识别了约15亿个变异,包括SNP、Indel和SV。与传统imputed阵列和WES相比,WGS使人类遗传变异的发现提升了18.8倍和40倍以上。该研究还证实了WGS发现的多个常见变异关联,这些关联在传统imputed阵列数据中曾被遗漏,包括与甲状腺功能减退风险及“其他白内障”(即眼部疾病)相关的基因位点。
SNP和Indel
研究团队使用GraphTyper调用了1,037,556,156个SNP和101,188,713个Indel(图2a)。几乎所有在WES数据集中发现的变异都可以在WGS数据集中找到。使用GraphTyper在至少1个个体中识别的变异数量是WES识别变异数量的42倍。在WES数据集中,外显子中转录但未翻译的变异被遗漏;WES数据集中分别缺失了69.2%和89.9%的5’和3’未翻译区域(UTR)变异。
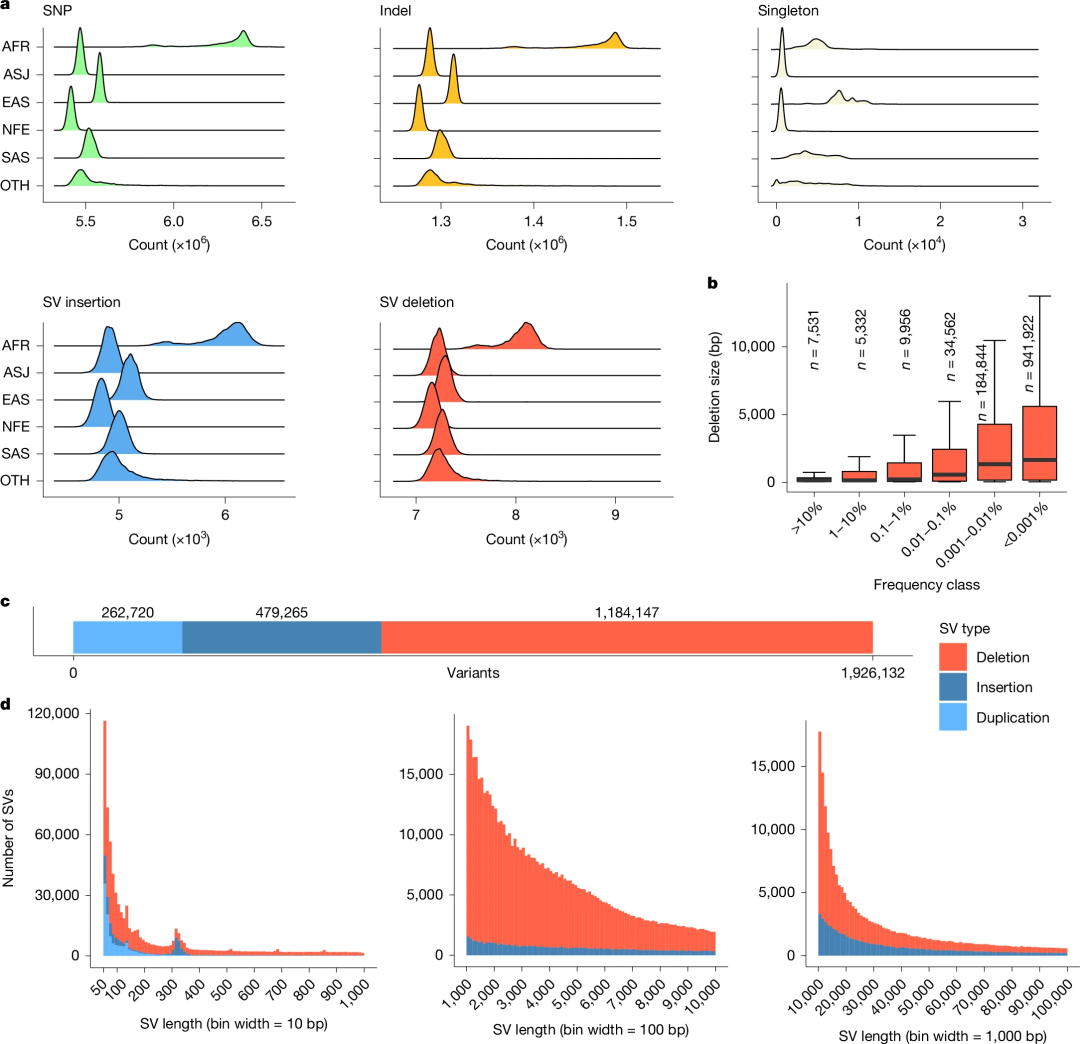
图2.变异检测。
通过对样本进行随机降采样,研究团队分析了UKB DRAGEN聚合变异数据集中,随着样本数量增加的变异数量。对于常见的变异(>0.1 %),没有观察到变异数量随着样本量的增加而增加,但是最罕见变异(≤0.001%)数量随着样本量的增加而大幅增加。该结果支持了进行大规模测序以发现新的和高影响的罕见变异的价值。
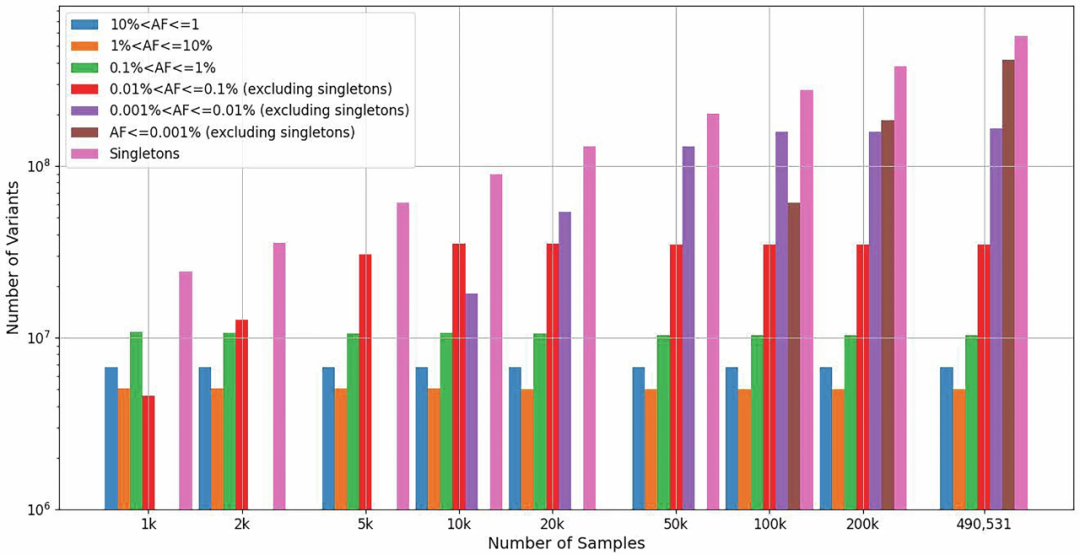
图3.样本量对变异数量的影响。
SV
研究团队使用GraphTyper对2739152例SV进行基因分型,其中70.3%被认为是可靠的,平均每个个体确定了13102个可靠的SV;7,340个缺失和5,762个插入或重复(图2b)。SV被定义为至少50bp的变异,其大小分布显示向短变异倾斜(图2d)。虽然SV的数量远小于SNP和Indel的数量,但每个单倍体基因组平均受影响的碱基对数量(3.6 Mb)与SNP(2.9 Mb)和Indel(1.5 Mb)相当。大多数SV都非常罕见,且罕见变异通常比常见变异长(图2b)。
此外,不同个体被调用的变异数量因人群而异,其中AFR队列个体被调用的变异数量最多,其次是EAS、SAS、ASJ,最后是NFE队列。与当前参考基因组相比,NFE队列中个体被调用的变异数量最少。
表型关联研究
研究团队整合了大多数UKB参与者可用的深层表型数据,并对通过电子健康记录和分子及物理表型捕获的选定疾病结果进行了遗传关联分析,包括全基因组关联分析(GWAS)、多祖先meta-GWAS、WGS中的功能丧失变异(LoF)、WGS和WES相关的罕见编码变异关联研究、UTR罕见变异PheWAS、SV的表型效应。
研究证实了WGS发现的多个常见变异关联,这些关联在传统imputed阵列数据中曾被遗漏,包括与甲状腺功能减退风险及眼部疾病相关的基因位点。同时,该研究通过分析不同祖先群体的基因组关联性及跨祖先关联,结合丰富的表型数据,揭示了新的遗传学和临床洞见。虽然疾病特征相关的大多数关联主要出现在欧洲裔人群中(其中93.5%为非芬兰欧洲裔,其余31,785例来自其他大陆族群),但非洲和亚洲裔人群也显示出显著或全新的信号。
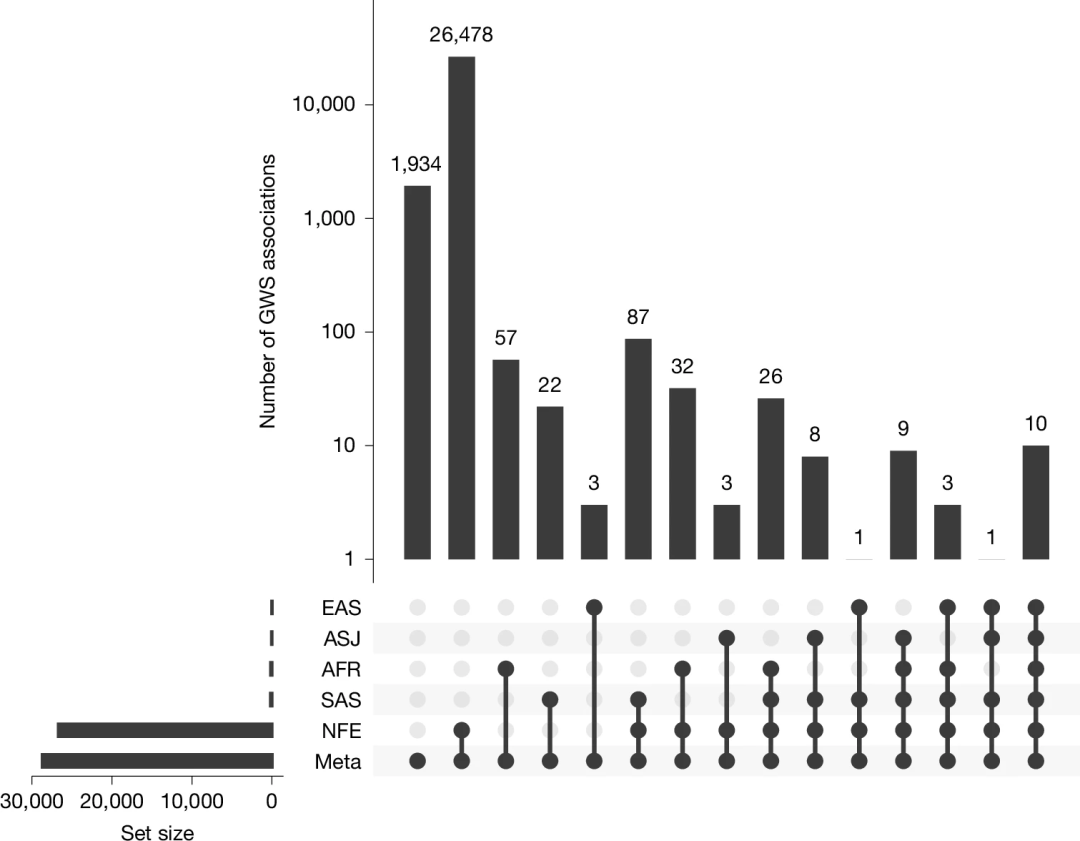
图4.全基因组显著关联在祖先群体中的分布图。祖先标签按每组GWS关联的数量进行排序:Meta、NFE、SAS、AFR、ASJ和EAS。
将UKB WGS与WES数据集进行比较,在同一组45万名参与者中,WGS和WES中≥1名携带者中约有16,000个常染色体基因携带pLoF、P或LP变异。但WGS能够在更多个体中鉴定出更多携带pLoF、P或LP变异的基因,为评估LoF杂合携带者甚至人类基因敲除中的基因靶标提供了更多的机会。
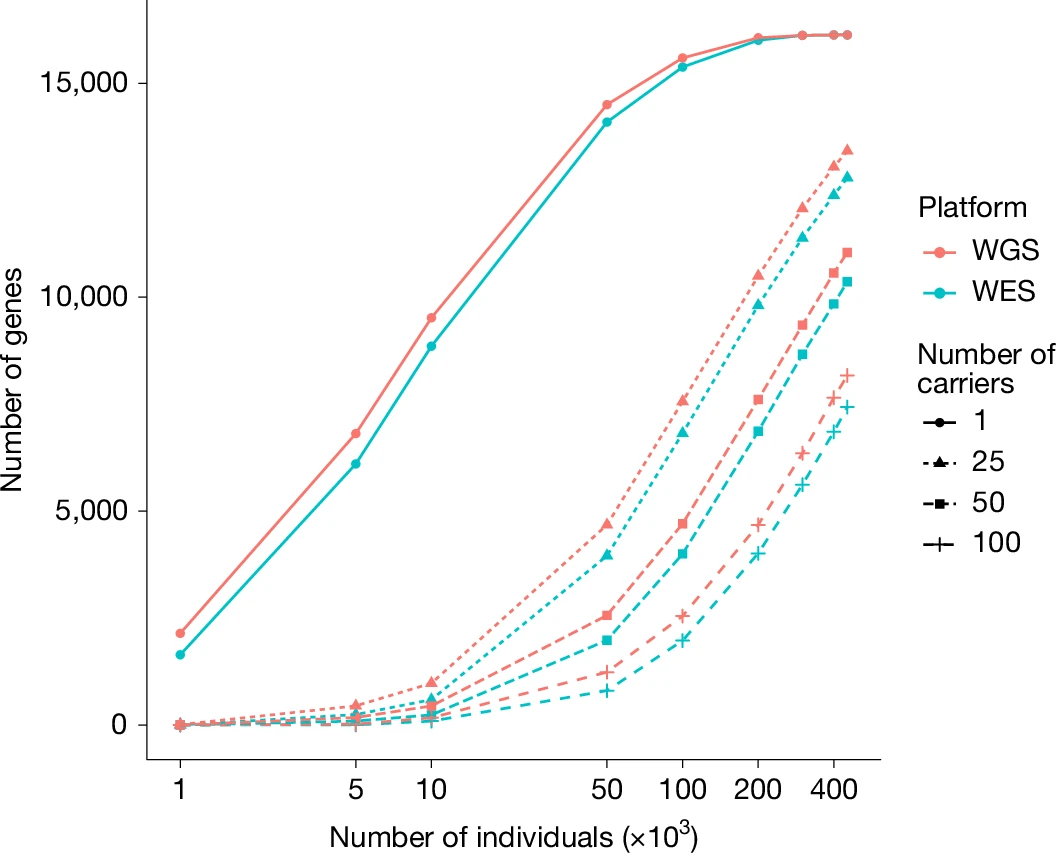
图5. WGS和WES中,携带杂合pLoF、P或LP变异受试者中的基因数量。
结 语
UKB WGS项目为探索人类遗传变异及其在疾病研究中的应用提供了一个开创性的机会。利用这个大规模、高度表型化的WGS数据集,研究人员可以深入探究非编码变异对健康与疾病的影响机制,能够精准定位具有特定致病遗传基础的患者群体、验证药物靶点、评估安全性风险、把握药物再定位机遇,并解答药物研发领域的其他关键问题。该研究将为罕见非编码变异对人类的影响提供必要的见解,并将促进未来十年人类遗传学向治疗的转化。
原文信息:
The UK Biobank Whole-Genome Sequencing Consortium. Whole-genome sequencing of 490,640 UK Biobank participants. Nature (2025). https://doi.org/10.1038/s41586-025-09272-9
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)