首页 > 医疗资讯/ 正文

【背景介绍】
空间转录组技术作为2020年《Nature Methods》评选的年度方法,开启了在组织切片中同时探测空间位置信息与基因表达水平的全新篇章。该技术能够让研究者观察组织内部各类细胞在空间上的分布状态,并揭示细胞之间在微环境中的相互作用,极大地丰富了我们对组织结构、细胞功能及疾病发生机制的理解。早期的空间转录组技术(如Spatial Transcriptomics)通过将组织切片铺设在带有DNA寡聚探针的芯片上,使RNA能够根据其空间位置被捕获并进行测序。然而,这些技术的探针直径约为100微米,约同时覆盖10至40个细胞,导致其仅能提供粗粒度的转录图谱,远不能满足研究者对于单细胞精度的需求。
随后,随着实验技术的发展,Slide-seq等平台将探针尺寸缩小至10微米,逐步接近单细胞分辨率。2022年问世的Stereo-seq更是将分辨率推向极致,探针直径达到220纳米,中心间距500纳米,实现了亚细胞级别的捕获能力。不仅如此,Stereo-seq还具备13.2厘米 × 13.2厘米的大视野测序能力,使得研究者可以覆盖整个小鼠胚胎等大尺度组织切片。可以说,空间转录组技术已步入高分辨率、高通量的新时代。然而,分辨率的提升也带来了全新的分析挑战——如何从如此精细的空间数据中准确地重建单细胞边界和类型,成为新的研究瓶颈。
传统空间转录组的计算任务大多集中在“去卷积”——即从低分辨率spot中推断混合的细胞类型。而随着亚细胞分辨率数据的出现,问题发生了根本转变:不再是从一个大spot中拆分多个细胞,而是要将大量亚细胞级别的spot聚合为完整的单细胞。现有主流方案大多采用“Bin X”策略,即将若干个探针点按照固定网格组合成一个虚拟的细胞,这些网格大小大致等同于目标组织中平均细胞的尺寸。这种方法虽操作简便,但严重偏离真实细胞的形态和位置。不同细胞间可能拥有截然不同的形状、大小及分布密度,使用统一网格划分会导致“伪细胞”的表达特征,进而影响后续的细胞类型识别、组织结构还原及功能分析。
近期,沙特阿卜杜拉国王科技大学高欣教授/南方科技大学胡宇慧副教授等人提出了STP方法,通过整合亚细胞分辨率的空间转录组数据与细胞核染色图像,实现了高精度的单细胞划分。相比传统方法,STP能够重建真实的细胞边界,揭示组织中的空间结构与稀有细胞类型。相关工作以“STP: single-cell partition for subcellular spatially-resolved transcriptomics”为题发表在Nature Communications。
这项研究中,STP致力于精确还原真实细胞边界。其核心思想是结合空间转录组数据与配套的细胞核染色图像。STP首先使用深度学习模型对染色图像中的细胞核进行实例分割,提取每个细胞核的掩码信息;随后将这些掩码精确映射至空间转录组的spot坐标系中;最后,借助模拟退火算法(Simulated Annealing)启发式策略,从核向周围spot扩展,直到形成完整的细胞区域。该策略在优化一个能量函数的同时充分考虑了细胞形态、表达相似性及空间约束,是首个融合图像和亚细胞空间转录组数据,且能支持大规模组织切片精确划分的计算方法。
【文章要点】
STP方法的核心优势在于其结合图像与空间转录组数据的多模态分析流程,主要包含四步(1)细胞核分割;(2)空间映射;(3)模拟退火扩展;(4)下游注释分析。首先,对染色图像进行增强和阈值去噪,再通过深度学习模型完成实例级细胞核分割。接着,将核掩码精确映射到空间转录组切片,建立图像与表达数据的空间对应关系。
第三步,STP使用模拟退火启发式策略扩展每个细胞边界。在每轮迭代中,从外围候选spot中选择扩展点,依据细胞形态、邻近表达相似性和空间布局进行能量函数评估。即使当前扩展方案效果略差,仍有概率被接受,以跳出局部最优并获得全局更优结构。最终,每个核被扩展为真实形态的细胞。
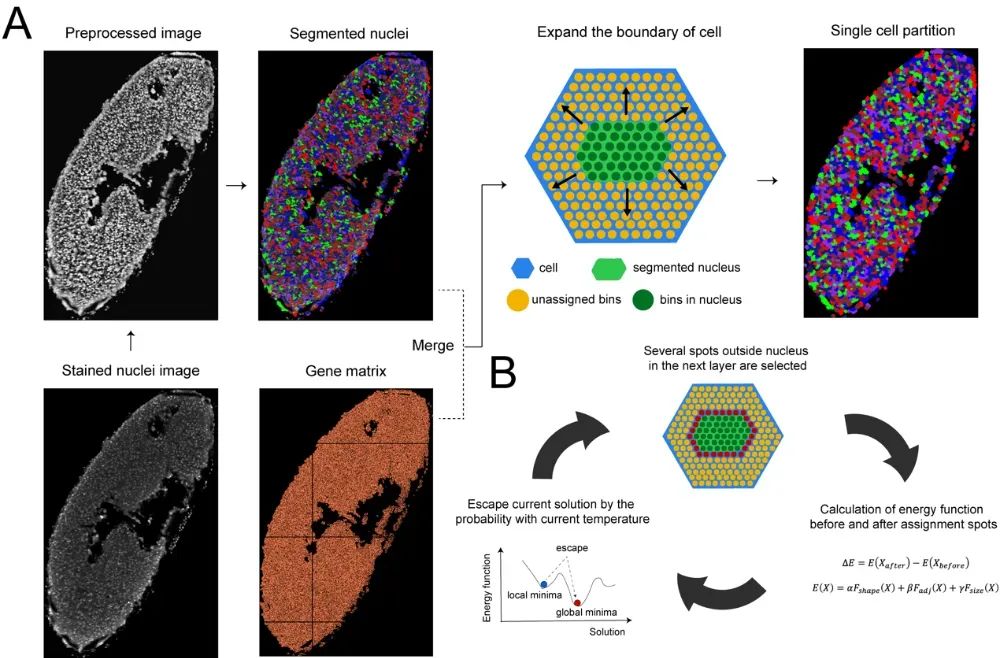
图1:STP的流程。
本文通过果蝇胚胎和小鼠胚胎两个经典模型系统对STP方法进行了系统评估。首先在果蝇模型中,研究者收集了两个发育阶段的胚胎样本(4-8小时与8-10小时),分别经冷冻切片与Stereo-seq测序,并采用ssDNA进行核染色。每张切片的空间转录组数据点数超过十万,配套的染色图像可提供核级形态参考。在这些样本中,STP首先分割出每个细胞核,然后对其进行空间映射与边界扩展操作。实验发现,STP分割出的每个细胞包含约100–200个spot,显著提高了空间组织信息的保真度。
进一步分析显示,STP划分出的细胞群在组织切片中呈现出良好的空间连续性,能够精确还原神经系统、胚外组织、表皮等多个细胞类型。作者通过标记基因表达热图与空间定位图进行对比,发现STP所识别出的细胞类型与标记基因在空间上的一致性显著高于传统核分割方法。此外,通过Point-Biserial相关系数(PBCC)等定量指标,STP在大多数切片中展现出更高的表达一致性。作者还通过Harmony消除不同切片间的批次效应,并使用PASTE算法完成三维重构,首次在果蝇模型中构建了连续的3D单细胞类型空间结构。
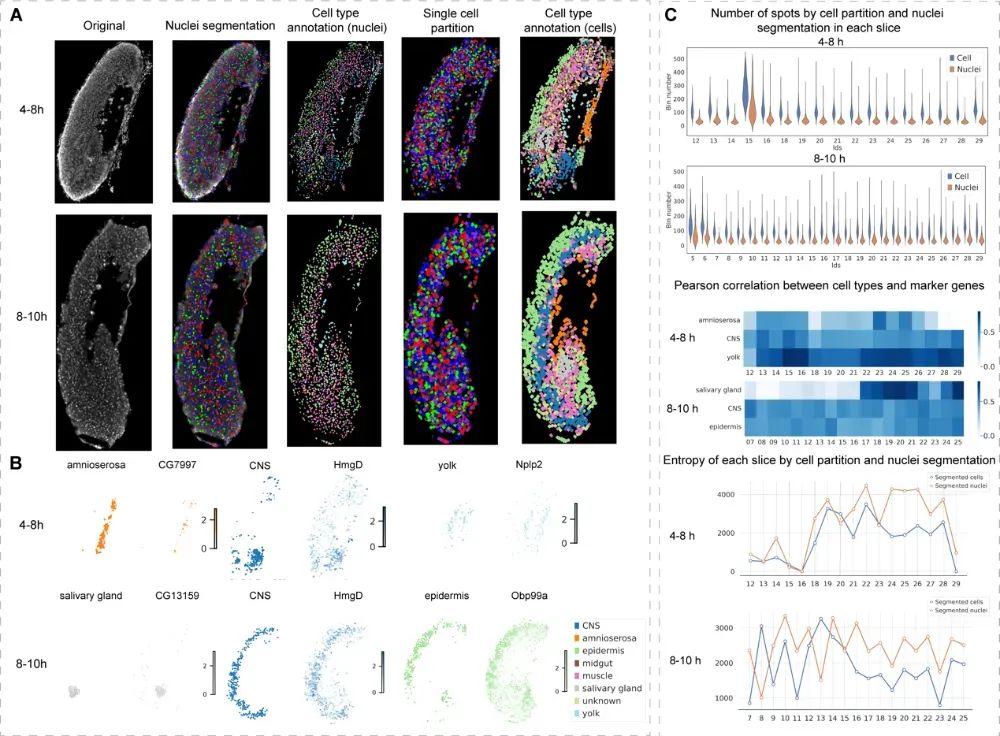
图2:果蝇胚胎数据的单细胞分割。
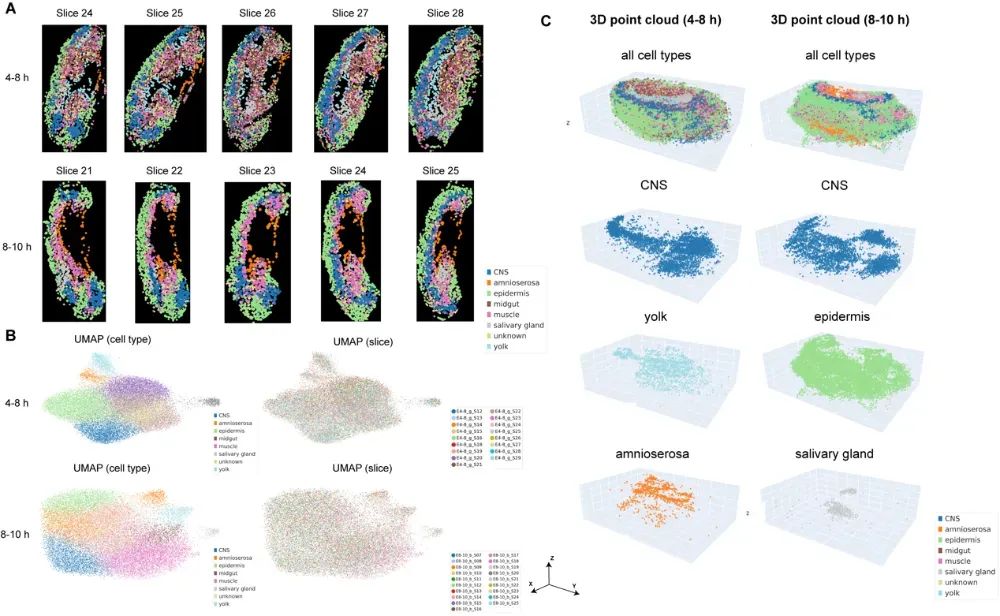
图3:在2D和3D层面上的不同细胞类型的连续空间分布。
在小鼠胚胎E14.5阶段的实验中,STP被应用于一个拥有7700万个spot的大视野切片,检测到近40万个单细胞,覆盖约2.7万个基因。STP不仅能够精确识别大脑、软骨原基、放射状胶质等组织类型,还显著优于KNN、Voronoi、Watershed等传统图像分割算法,在多个指标(PBCC、PCC、MSE、细胞尺寸分布等)上取得领先表现,尤其在识别稀有或结构复杂的细胞群体方面展现出独特优势。
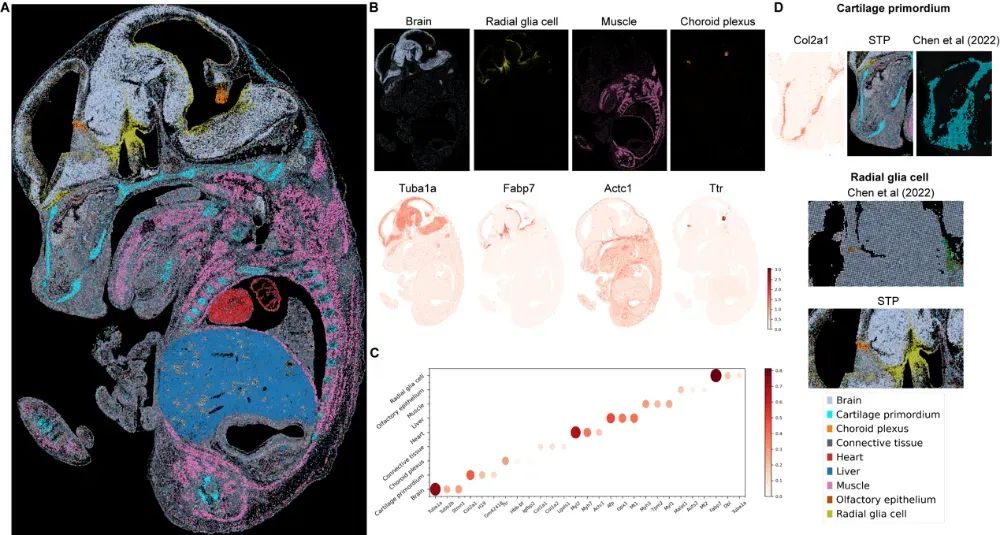
图4:小鼠胚胎数据的单细胞分割。
【总结与展望】
STP方法以其创新性在空间转录组单细胞划分领域开辟了新路径。通过整合亚细胞分辨率的空间表达信息与核染图像,STP构建了一个兼具精度与可扩展性的细胞分割框架,在多样本、多组织、多物种的实验中展现出良好的泛化能力。相比于传统“Bin X”策略,STP可有效避免形态失真、类型混淆等问题,尤其在识别稀有细胞类型、重建三维组织结构及揭示发育动态方面具有显著优势。该方法在果蝇胚胎中重构的连续细胞图谱和在小鼠中识别出的罕见细胞类型,表明其在发育生物学、组织工程乃至疾病研究中具备广泛的应用潜力。
尽管STP表现出色,但仍存在改进空间。其性能依赖多个超参数,如初始温度T、能量函数构建方式等,对不同组织结构的适配性仍需优化。此外,其对数据质量较为敏感,染色图像中若出现过染或细胞密集交叠,可能影响核分割准确性。同时,当前工作主要以经验性指标评价STP优越性,未来可引入统计假设检验等更严格的量化分析手段以提升结果可信度。随着空间转录组和染色技术的进一步发展,未来STP有望被拓展为一套通用的亚细胞数据分析工具。通过该方法生成的高维空间表达图谱,也有望成为疾病诊断、生物标志物发现、个体化医疗等方向的重要信息源。
原文链接:
https://www.nature.com/articles/s41467-025-59782-3
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)