首页 > 医疗资讯/ 正文
传统肿瘤分类主要依据肿瘤的组织类型、细胞类型和生物学行为。随着分子生物学和基因测序等技术的发展,肿瘤分类模式也发生了转变。DNA甲基化是一种重要的表观遗传修饰,在基因表达和细胞分化调控中具有重要作用,与肿瘤等许多人类疾病有关。目前,基于DNA甲基化的肿瘤分类已经成为建立临床诊断和研究癌症分子分类的重要诊断工具。此外,脑肿瘤的综合组织-分子分类已被证明可以改进组织学诊断。
目前的DNA甲基化检测方法包括亚硫酸氢盐全基因组测序(WGBS)、靶向甲基化测序、微阵列、纳米孔测序等。但不同检测方法的基因组覆盖范围和深度需要不同的特异性分类分析方法,已有的机器学习算法常限于单平台数据或固定的甲基化特征空间,这导致平台间的不兼容。因此,我们迫切需要一种能够跨平台预测肿瘤分类的精确模型。
为此,德国柏林夏里特大学医学院研究团队提出了一种基于神经网络的机器学习框架crossNN,可以使用不同平台的不同表观基因组覆盖范围和测序深度的稀疏甲基化数据实现准确的即时肿瘤分类,其性能优于已有的深度学习模型和传统机器学习模型,并且可解释。研究显示,基于crossNN训练的泛癌分类模型,可以区分人类所有器官部位的170多种肿瘤类型。在不同测序平台对5000多个肿瘤的验证显示,脑肿瘤和泛癌模型的精确度分别为99.1%和97.8%,表明该模型具有较高的稳健性和可扩展性。该研究成果已发表在Nature Cancer上。

crossNN模型开发与验证
crossNN模型架构依赖于感知器(perceptron)(图1),该感知器使用PyTorch实现为单层神经网络。网络结构包括输入层和输出层,两层完全连接,无偏置,意味着该模型将捕获输入CpG位点和甲基化分类(MC)之间的线性关系。
在模型训练中,研究团队使用了海德堡脑肿瘤分类器v11b4参考数据集。该数据集由来自82种肿瘤类型和亚型以及9种非肿瘤对照的2,801个样本的甲基化谱组成(微阵列生成),训练数据集的特征空间是固定的。crossNN模型使用随机和重复屏蔽的输入数据进行训练,以实现在不同平台上对不同或稀疏表观基因组覆盖的甲基化组分析进行肿瘤分类。
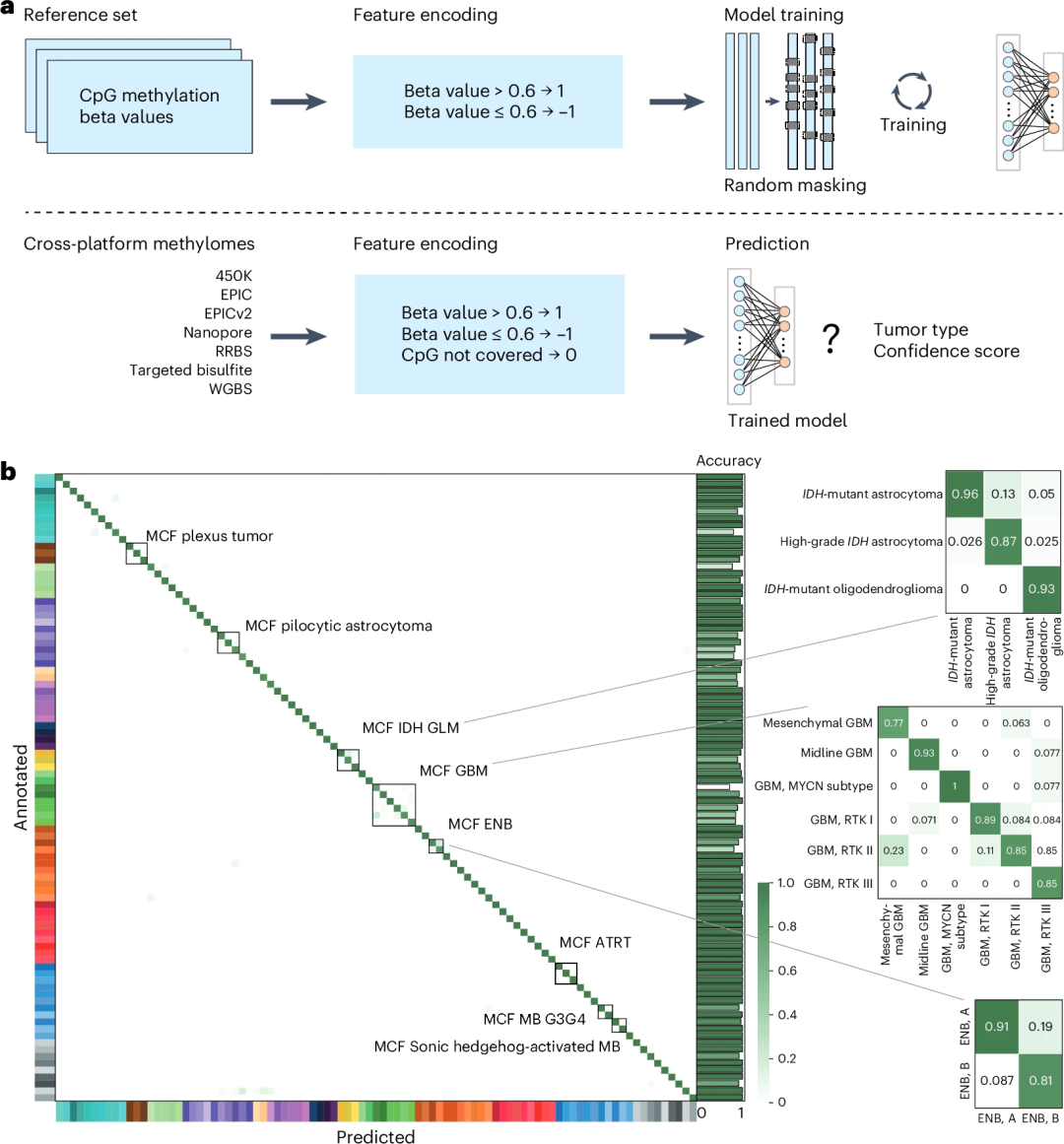
图1.crossNN模型架构、训练和交叉验证。
研究人员对crossNN模型进行了性能评价。(图1)首先,在训练数据集中使用5倍交叉验证评估模型的性能。在MC水平上,该模型的总体准确率为96.11±0.86%;在甲基化分类家族(MCF)水平上,预测准确率达到99.07±0.21%。此外,研究人员以不同采样率(0.5%到100%)进行二次采样,测试该模型在不同CpG位点覆盖率下的性能,发现crossNN模型表现出稳健的性能。在五倍交叉验证中,在0.5%到75%的不同采样率下,crossNN模型仍具有很高的平均准确率,能够在几秒钟内进行预测,优于ad-hoc RFs模型。
随后,研究团队在不同微阵列和测序平台上生成的独立队列中验证了最终crossNN模型。研究收集了2090个患者样本,这些样本来自Illumina 450K、EPIC和EPICv2微阵列,纳米孔低通量WGS,Illumina靶向甲基化测序和Illumina WGBS。该数据集涵盖了62种不同脑肿瘤类型,涉及训练集中82种甲基化分类中的67种。crossNN模型在MC水平和MCF水平的总体精度分别为0.98和0.99。
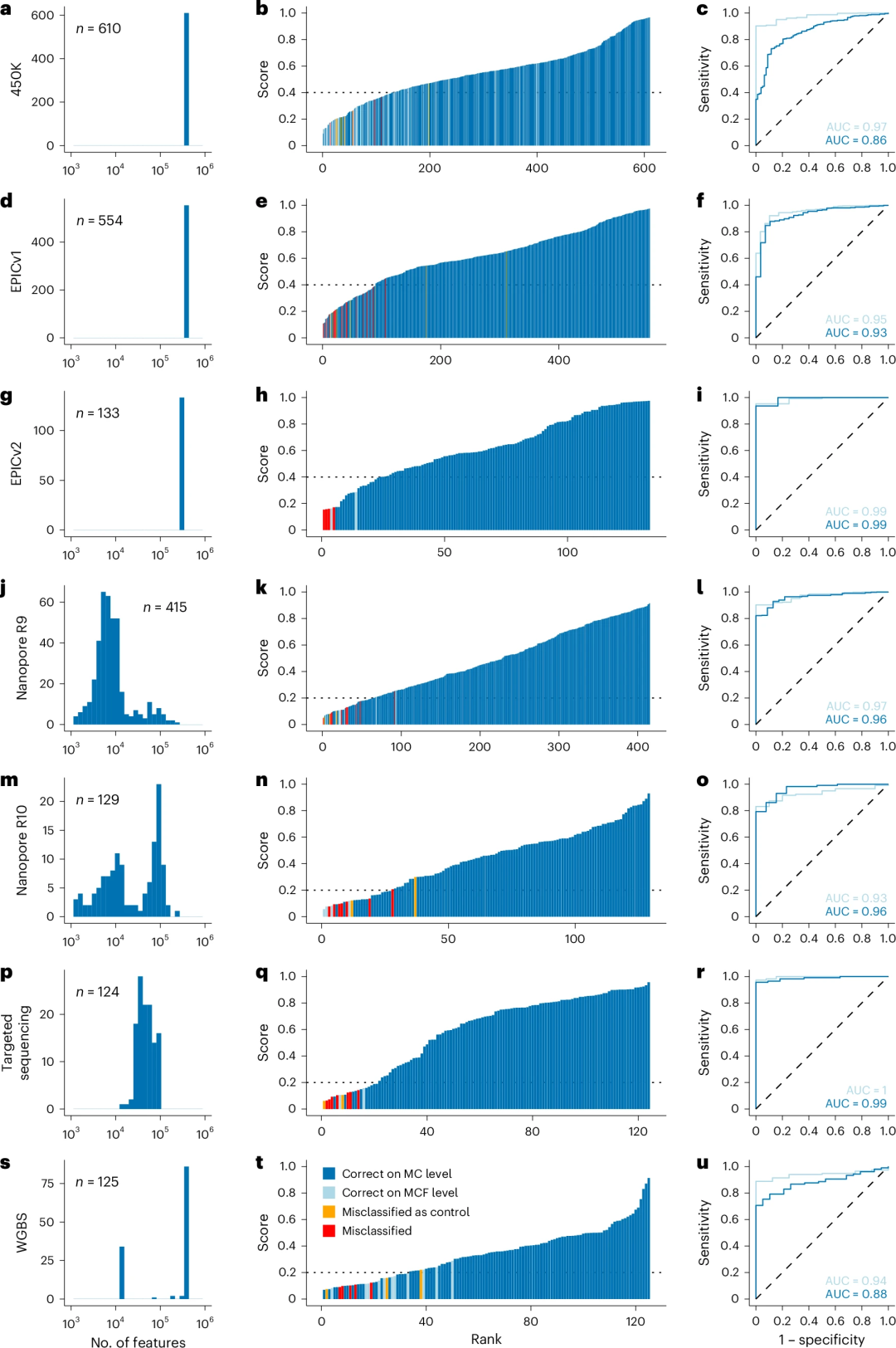
图2.验证队列中的分类结果
研究团队将crossNN模型、ad-hoc RF方法和其它已发表深度神经网络方法(Sturgeon DNN)进行了比较。数据显示,crossNN模型在总体精度方面与ad-hoc RF和Sturgeon DNN相当,在预测分数的ROC特征方面,特别是精度方面优于两种方法。此外,在可解释性分析中,crossNN模型揭示的特征重要性揭示了个体标记基因的功能重要性,并可以量化表观遗传修饰在基因结构中的位置重要性。因此,crossNN模型架构使用表观基因组特征进行分类,这些特征可以单独与转录调控和细胞信号传导联系起来,使模型完全可解释。
crossNN泛癌模型
研究团队集合了一个泛癌参考数据集来训练crossNN泛癌模型。训练数据集由公开的、来自大多数器官部位的178种肿瘤类型的8,382例病例组成。内部验证和五倍内部交叉验证结果显示,crossNN泛癌模型的MC水平和MCF水平的总体准确率分别为94.82±0.06%和97.61±0.05%。在5379例未知病例验证中,该模型MC和MCF水平的总体准确率分别为0.83和0.88。在所有主要肿瘤类型中,crossNN泛癌模型的准确率都很高;与原发性脑肿瘤、肉瘤和淋巴细胞恶性肿瘤相比,癌症亚型队列的准确性较低。此外,研究引入了一个“鳞状细胞癌超家族”MCF,以降低可能出现的错误分类,例如乳头状和透明细胞肾癌。
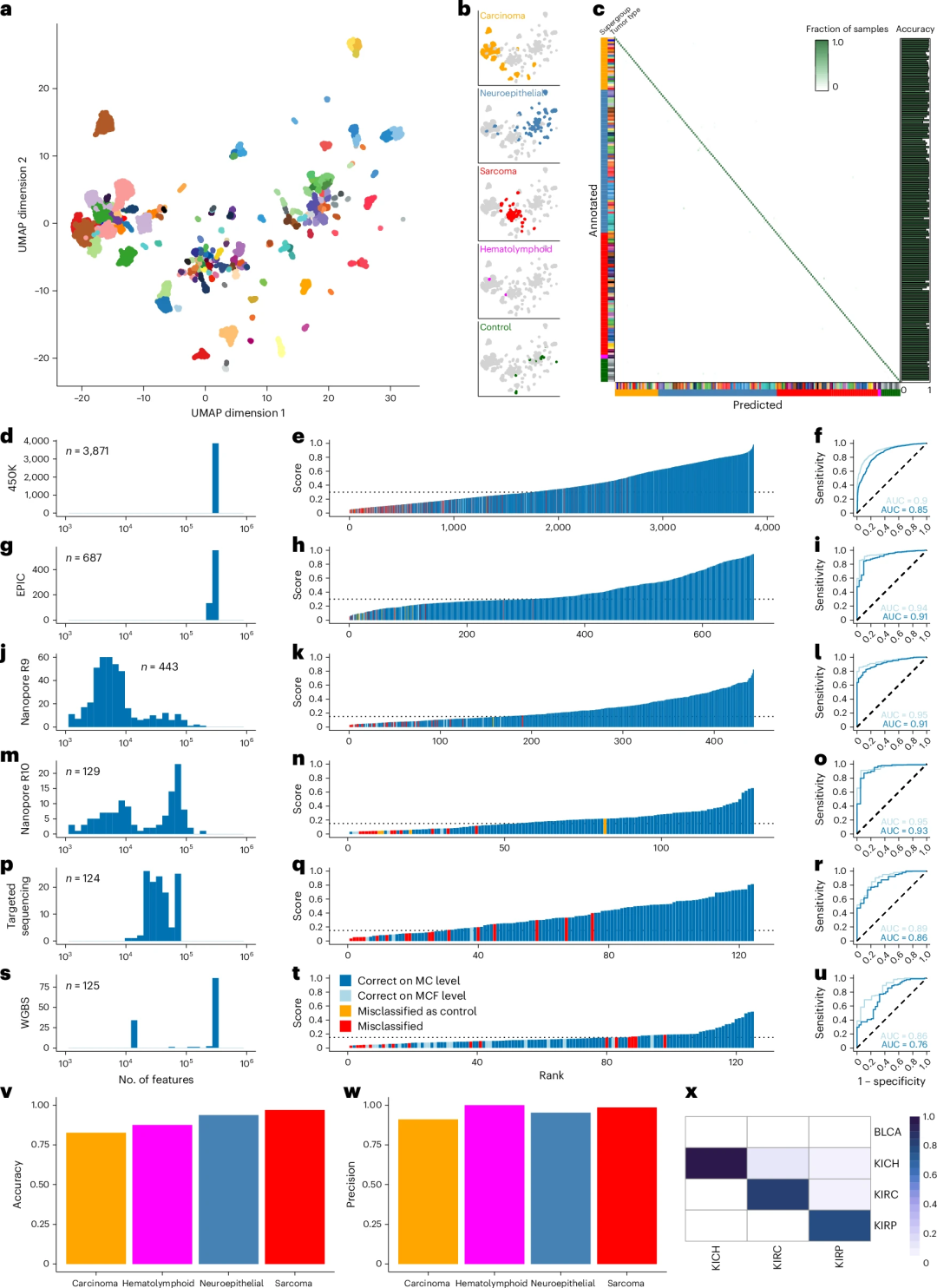
图3.crossNN泛癌模型的验证
结 语
该研究提出的跨平台、基于DNA甲基化的机器学习框架crossNN,使开发快速、有弹性、可解释和准确的癌症诊断成为可能。crossNN简单且可解释,可以使用来自不同平台的DNA甲基化谱对实体瘤进行即时分类,并在精度、简便性以及计算需求方面优于其他机器学习模型。该模型的鲁棒性和可扩展性在低通量纳米孔WGS、WGBS、靶向甲基测序和微阵列脑肿瘤队列中得以验证,crossNN泛癌模型进一步证明了其具有高度可扩展性。同时,轻量级可扩展架构允许crossNN快速再训练和交叉验证,有助于快速适应新的癌症参考图谱。
文章通讯作者、德国柏林夏里特大学医学院Philipp Euskirchen博士表示:“成千上万的表观遗传修饰充当了单个基因片段的开关,它们形成了一个独特的、不会弄错的指纹。在肿瘤细胞中,表观遗传信息以特有的方式改变,根据这些信息,我们可以区分肿瘤并进行分类。新开发的模型可以在99.1%的情况下非常精确地诊断脑瘤,比迄今为止有效的AI解决方案更准确。该模型可以区分来自所有器官的170多种肿瘤类型,同时达到97.8%的准确率。这意味着它可以用于所有器官的癌症类型,除了相对罕见的脑肿瘤。”
crossNN模型的即时预测极大地改善了时间紧迫的临床应用,例如术中诊断。此外,crossNN将DNA甲基化分类的应用范围扩展到脑肿瘤之外,并将在未知原发癌症的诊断工作中有重要作用。重要的是,该模型与EPICv2微阵列平台兼容。研究团队开发了一个直观的基于web的图形用户界面,允许用户上传甲基化数据并即时预测实体瘤(https://crossnn.charite.de)。此外,模型和源代码可用于本地部署和与机构工作流集成(https://gitlab.com/euskirchen-lab/crossnn)。
未来,该研究团队与德国癌症联盟合作,希望在德国所有八个联盟地点进行临床试验,以进一步测试crossNN的准确性。
论文原文:
Yuan, D., Jugas, R., Pokorna, P. et al. crossNN is an explainable framework for cross-platform DNA methylation-based classification of tumors. Nat Cancer (2025). https://doi.org/10.1038/s43018-025-00976-5
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)