首页 > 医疗资讯/ 正文
人类微生物组由胃肠道中大量多样的微生物组成,与各种人类健康和疾病相关状况有着重要联系。微生物群落的行为和生物效应不仅取决于其物种组成和多样性,还取决于每种微生物中出现的细胞状态。细菌细胞状态在很大程度上受到群落中包括的每种微生物的基因转录活性的影响。例如,以前的研究报道,大肠杆菌在抗生素治疗后具有不同的持久性表型,这是由基因转录活性的异质性引起的。亚群内细菌转录状态的变化会导致抗生素耐药性和代谢能力等基本特征的差异,这可能对人类健康产生重大影响。因此,鉴于细菌种群之间众所周知的功能异质性,有必要采用进一步的方法来实现宿主相关微生物的完整功能表征。
过去十年中,单细胞 RNA 测序的进步及其在哺乳动物系统中的成功应用揭示了细胞类型和状态的巨大转录异质性,并改变了多个方面的生物学研究。值得注意的是,几种细菌 scRNA-seq 方法、PETRI-seq、MATQ-seq、par-seqFISH、ProBac-seq、BacDrop、smRandom-seq是最近开发的。然而,在几个主要限制中,目前的微生物技术仅限于特征明确的分类群,仍然不适用于临床和生理学相关的人类微生物组样本。因此,使用现有方法的研究仅关注少数特征明确的细菌的种群异质性,例如人工实验室生成的培养混合物。因此,迫切需要一种针对世界上表征较差和复杂的微生物群落的单微生物 RNA 测序(smRNA-seq)技术。
3月8日,浙江大学王永成团队在Protein & Cell(IF=13.6)在线发表了题为“High-throughput single-microbe RNA sequencing reveals adaptive state heterogeneity and host-phage activity associations in human gut microbiome”的研究,这项研究介绍了 smRandom-seq2,这是一种高度适应复杂微生物群落的高通量和高分辨率 smRNA-seq 方法。通过分析从健康人类受试者收集的 4 份粪便样本,我们成功地从肠道样本中获得了 29,742 个单一微生物条形码。进一步开发了复杂微生物群落中微生物注释和细菌-噬菌体转录活性的分析管道。总而言之,研究证明了 smRandom-seq2 是一种用于研究单个微生物转录活性的新型测序技术,可以深入了解人类肠道微生物组中细菌和噬菌体之间的功能异质性和相互作用。

该研究开发了一种针对复杂微生物群落的基于液滴的高通量和高灵敏度 smRNA-seq 方法 (smRandom-seq2)。
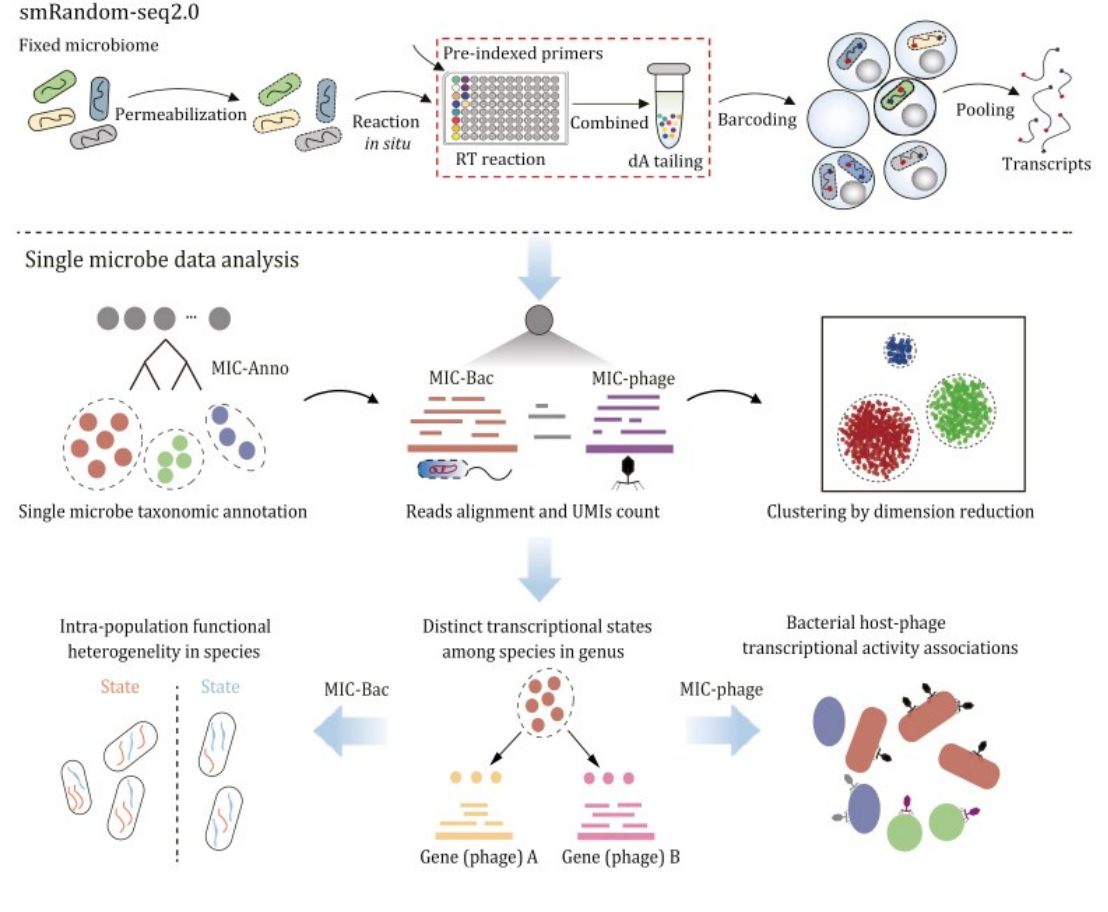
图1.smRandom-seq2 和数据分析概述

图2.人类肠道微生物组中的细菌基因表达景观
与现有的 smRNA-seq 方法相比,smRandom-seq2 克服了真实微生物组样本中普遍存在的高度物种多样性和异质性挑战。smRandom-seq2 进展背后的关键是:(1)在 smRandom-seq2 中,随机引物的新设计显着提高了所有细菌物种的逆转录效率。(2)在 smRandom-seq2 中,基于预索引的实验过程显着减少了复杂微生物群落的单个微生物测序中的交叉污染。在 GAT 随机引物上引入了 pre-index 设计,以减少细菌聚集引起的交叉污染。基于预指数策略,即使它们包含在同一个液滴中,也可以区分两种细菌。结果表明,在 smRandom-seq2 中,条形码的平均物种纯度高于 95%,这是其他方法在复杂微生物组上很难实现的。(3)以前的单微生物测序平台,如 BacDrop 和其他基于液滴的单细菌测序技术,是基于 10× Genomics 单细胞条形码平台开发的,该平台主要是为真核生物单细胞开发的,由于微生物体积小得多,并且微生物组样品中存在大量杂质,因此对单微生物条形码效率不高。在 smRandom-seq2 中,开发了一种高条形码效率的液滴平台,特别适用于复杂的微生物组单微生物测序。(4)在smRandom-seq2 中,还开发了一种新的计算管道,用于复杂微生物群落中的单个微生物分析。这种计算方法首次解决了多物种单细胞分析的问题,极适用于复杂微生物群落中的单微生物测序。通过通过 smRandom-seq2 汇集 smRNA-seq 数据,能够获得每种微生物的准确分类信息,并进一步进行细菌和宿主噬菌体关联分析。由于本研究的上述改进,能够在人类肠道微生物组样本中进行单一微生物测序。
事实上,smRandom-seq2 在人类粪便样本上的应用表明,smRandom-seq2 可以灵敏地剖析存在于复杂微生物群落中的功能异质性,并在属和物种水平上识别宿主-噬菌体活性关系。smRandom-seq2 还可以同时从人类肠道中识别数百种细菌种类,突出了它在物种覆盖的深度和范围以及效率方面的独特优势。通过在人类粪便样本中应用 smRandom-seq2,生成了数以万计的人类单一微生物,研究界可以轻松访问和利用这些微生物。总之,该研究结果支持 smRandom-seq2 具有用于各种生物医学甚至临床研究的潜力,从了解微生物组功能变化和细菌动态通讯,到细菌代谢网络和宿主-微生物组相互作用等。此外,smRandom-seq2 能够以单个微生物分辨率捕获微生物组转录状态,为了解复杂的微生物群落及其对人类健康的功能影响提供了新的途径。
基于对单个微生物的系统噬菌体分析,发现大多数噬菌体与活性转录的原噬菌体有关。经过进一步调查,发现绝大多数这些噬菌体比对的读数 (超过 85%) 可以定位到细菌基因组。这表明大多数病毒处于原噬菌体状态,原噬菌体的转录活性与人肠道微生物组中细菌的适应状态显著相关。在确定的 373 种可靠的预测宿主-噬菌体关系中,每种噬菌体物种基本上都与特定的细菌属有关。只有 8 种噬菌体表现出与多个细菌属的关联。其中许多噬菌体是不可培养的,因此数据集中很大一部分缺乏分类学和基因组信息。在肠道微生物组样本中鉴定的 373 种宿主-噬菌体关系中,鉴定了 18 种涉及宿主和噬菌体的关系,并具有明确识别的分类信息。还发现这些原噬菌体相关的细菌功能基因主要参与关键功能途径,特别是精氨酸和色氨酸代谢。此外,一些功能基因表现出与细菌应激保护的相关性,表明它们在增强细菌与支持恢复力的原噬菌体共存方面的潜在作用。
总之,该研究建立了人类肠道微生物组的单细胞水平细菌转录景观,发现了 Prevotella 和 Roseburia 属物种之间不同的适应性反应状态。并在人类肠道微生物组中鉴定了数百种新的宿主-噬菌体转录活性关联。结果表明,smRandom-seq2 是一种高通量和高分辨率的 smRNA-seq 技术,高度适应现实世界中的复杂微生物群落,并为理解人类微生物组带来新的前景。
原始出处:
Shen Y, Qian Q, Ding L, Qu W, Zhang T, Song M, Huang Y, Wang M, Xu Z, Chen J, Dong L, Chen H, Shen E, Zheng S, Chen Y, Liu J, Fan L, Wang Y. High-throughput single-microbe RNA sequencing reveals adaptive state heterogeneity and host-phage activity associations in human gut microbiome. Protein Cell. 2025 Mar 8;16(3):211-226. doi: 10.1093/procel/pwae027. PMID: 38779805; PMCID: PMC11891138.
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)