首页 > 医疗资讯/ 正文
基因组重复元件约占人类基因组的一半以上,包括各种不同的元件,其在个体之间差异较大,可对基因组结构和功能产生关键影响。DNA重复元件往往会被忽视,所以也被称为“垃圾DNA”或“暗物质”。重复元件主要由串联重复和逆转录转座子组成,其中串联重复是以相对恒定的短序列为重复单位,首尾相接、串联连接形成的重复序列,又称卫星DNA;反转录转座子包括各种全基因组重复家族,如长散布核序列(LINEs)、短散布核序列(SINEs)、长末端重复(LTRs)和其他转座元件等。
重复元件中的基因变化是癌症和其他疾病的标志,但使用标准测序方法表征这些变化一直是个挑战。近年来,随着液体活检技术的快速发展,使得人们能够分析游离DNA(cfDNA)中的重复元件,但目前尚未有研究对不同人类癌症组织或cfDNA中的重复序元件概况进行全基因组的系统分析。
为应对上述挑战,约翰霍普金斯大学医学院的科研团队在Science Translational Medicine上发表了题为“Genome-wide repeat landscapes in cancer and cell-free DNA”的研究文章,介绍了新开发的一种名为ARTEMIS(Analysis of RepeaT EleMents in diSease)的全新kmer(短DNA序列)机器学习分析方法,可以用于研究癌症中的重复元件变化。利用该方法,研究团队分析了2,837个组织和血浆样本中的12亿个kmers,共发现有1,280种重复元件显示出肿瘤特异性变化,其中包括先前未知的元件;重复元件在驱动基因区域富集,其表达方式受结构变化和表观遗传状态的影响。
在交叉验证和外部验证的队列中,研究团队对检测到的早期肺癌或肝癌患者的cfDNA全基因组重复序列图谱和片段分布进行机器学习分析,发现这些重复序列图谱可用来无创地识别肿瘤的起源组织。以上分析揭示了人类癌症重复元件图谱的广泛变化,并提供了一种无创检测和表征癌症的方法,这可能有利于癌症患者的早期检测和疾病监测。

文章发表在Science Translational Medicine
研究团队开发的ARTEMIS是一种无比对的全基因组方法,用于短读长测序中的kmers从头检索,以识别基因组中的重复元件(图1)。研究团队首先对来自1,975名不同癌症类型患者的2,837个组织和血浆样本进行分析,包括肺癌、乳腺癌、结直肠癌、卵巢癌、肝癌、胃癌、头颈癌、膀胱癌、宫颈癌、甲状腺癌或前列腺癌,共确定了12亿个24bp kmers,涉及1,266种重复元件类型。进一步,研究团队使用ARTEMIS对上述kmers进行深入分析,有1280种重复元件在肿瘤中发生了变化,涵盖转座元件、SINE、卫星DNA、LTR、LINE和RNA元件等6个家族。
研究团队发现重复元件在人类癌症经常改变的基因中富集,这些基因包括在癌症中扩增、删除和重排的基因;在已知的736个癌症驱动基因中,有487个包含的重复kmers数量平均比预期高出15倍。此外,以上重复kmers在涉及细胞信号通路的基因中也显著增加,这些信号通路在癌症中通常失调。上述重复kmers定位的观察结果表明,在肿瘤发生过程中重复相关的基因组变化,可影响人类癌症致癌途径的关键基因的改变。
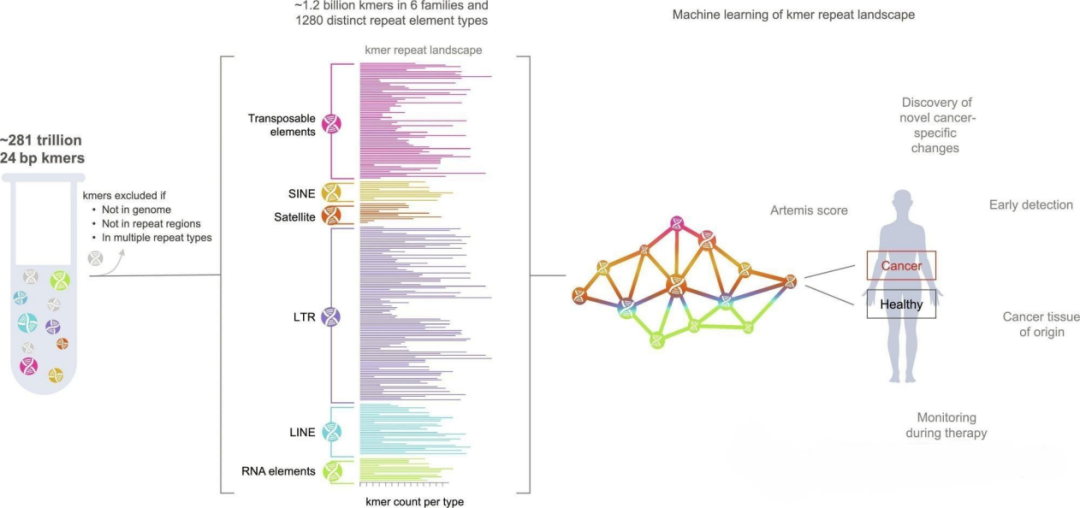
图1. ARTEMIS方法概述。该方法在癌症患者样本中发现了12亿个独特的kmers,涵盖1280个不同重复元件。
接下来,研究团队使用ARTEMIS分析了参与全基因组泛癌症分析(PCAWG)的525名不同癌症患者的肿瘤和匹配正常组织,以确定肿瘤中特定重复元件类型的全基因组kmer计数是否发生改变(图2)。kmers计数反映了肿瘤中全基因组染色体臂的增加或减少,与染色体不稳定性、基因组熵、杂合性丧失等全基因组结构变化具有相关性。结果显示,在30-60倍测序覆盖率下的每个样本中平均鉴定出224亿个总kmers,与1280种重复元件类型相对应。与匹配的正常组织相比,肿瘤中平均有807种个重复元件的kmer计数增加或减少;近三分之二的改变元件(1280种中的820种)在先前研究中未被观察到。
来自卫星、LINE和SINE的元件变化率最高,LTR内的元件、转座元件和RNA元件也经常观察到变化。此外,有近四分之一的元件来自LTR最大的重复亚家族,ERV1(内源性逆转录病毒1),其中约40%发生了改变。
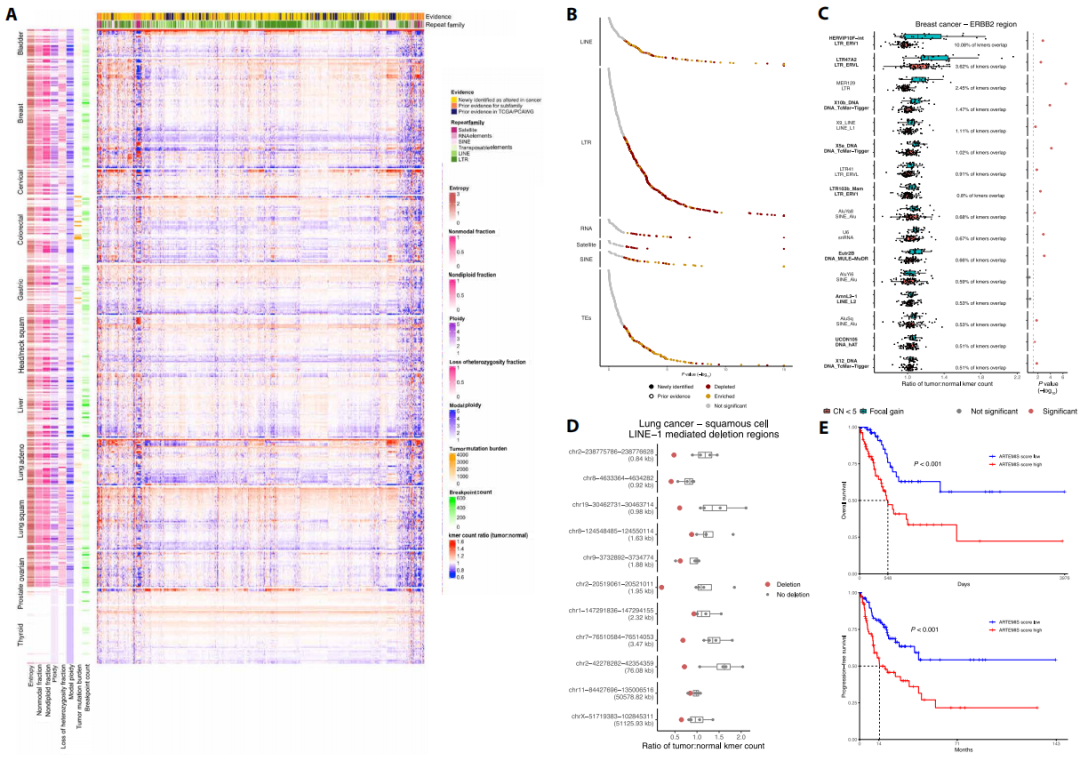
图2. kmers在人类癌症中的重复序列。
此外,研究团队使用机器学习模型为每个样本生成了ARTEMIS评分,以提供可预测癌症的全基因组重复元件变化总结。结果显示,ARTEMIS评分将525个PCAWG参与者的肿瘤与正常组织样本准确区分开,AUC为0.96;在所有癌症类型中,ARTEMIS评分增加均与患者的总生存期和无进展生存期缩短有关。上述结果表明,ARTEMIS评分捕获了重复序列的全基因组变化,癌症基因组中重复元件的重新激活和增加可能导致免疫反应增加或基因组不稳定,这两种机制都可能降低肿瘤细胞的适应性并改善患者的预后。
研究团队还分析了cfDNA中重复元件与基因组DNA中预期重复元件的差异(图3),发现全基因组染色质和表观遗传变化可能改变cfDNA片段在体液循环中的表现。随后,研究团队使用低覆盖率全基因组测序检测了cfDNA的重复元件。结果显示,cfDNA中的kmers重复元件在不同测序平台和实验批次中保持一致,且男性血浆中位于Y染色体上的卫星家族kmers计数显著高于女性。
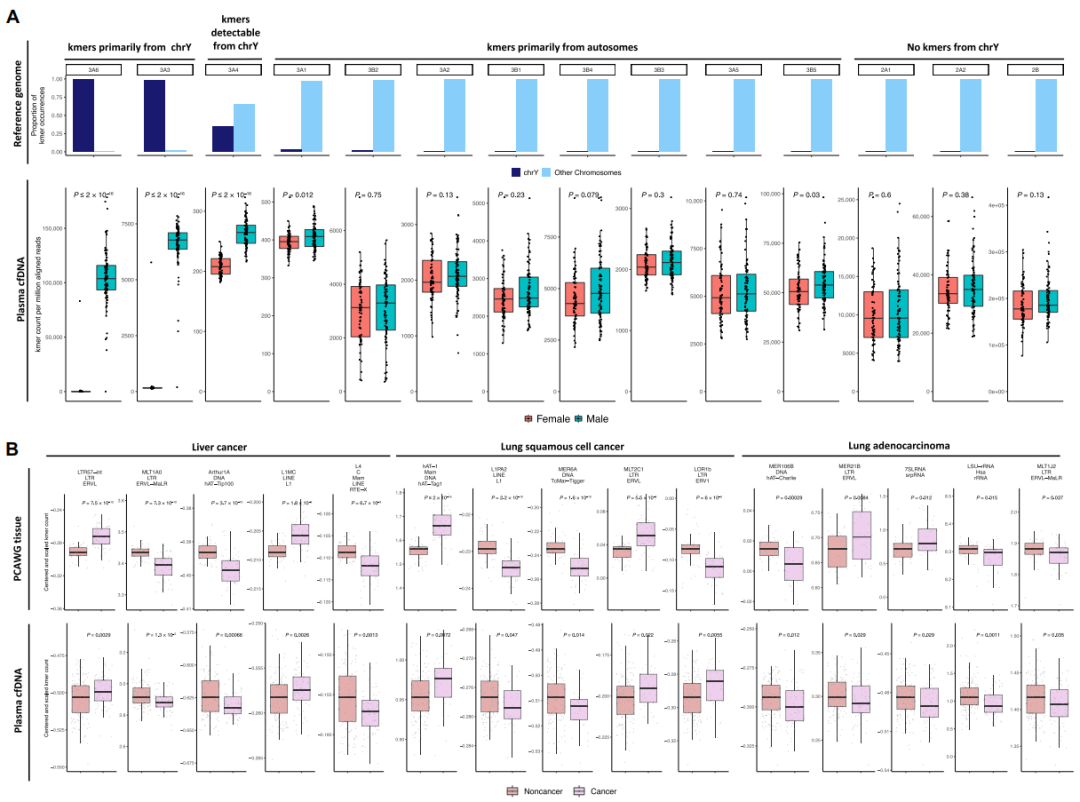
图3. kmers重复元件捕获血浆中肿瘤特异性的变化。
研究团队评估了ARTEMIS在癌症无创检测方面的潜力,将其应用于287名参与丹麦肺癌筛查研究(LUCAS)的肺癌和非肺癌患者血液样本中。结果显示,ARTEMIS可对肺癌患者进行分类,总AUC为0.82;当其与DELFI方法结合使用时,对肺癌患者分类的AUC为0.91。DELFI是由约翰·霍普金斯大学Velculescu团队开发的一种简易的血液检测方法,可通过cfDNA独特的片段化模式对癌症进行检测(点击查看更多相关内容)。在208名有肝癌风险的个体中,研究团队观察到了上述类似表现:ARTEMIS在肝硬化或病毒性肝炎中检测到肝癌个体时,AUC为0.87;与DELFI联合使用时,AUC增加到0.90。
研究团队还评估了肺癌或肝癌风险患者cfDNA中变化最大的重复元件的发生率,发现许多重复kmers在癌症患者血浆中增加或减少(图4)。全基因组模拟结果表明,cfDNA中的重复序列图谱可能受到肿瘤特异性表观基因组和基因组变化的影响。
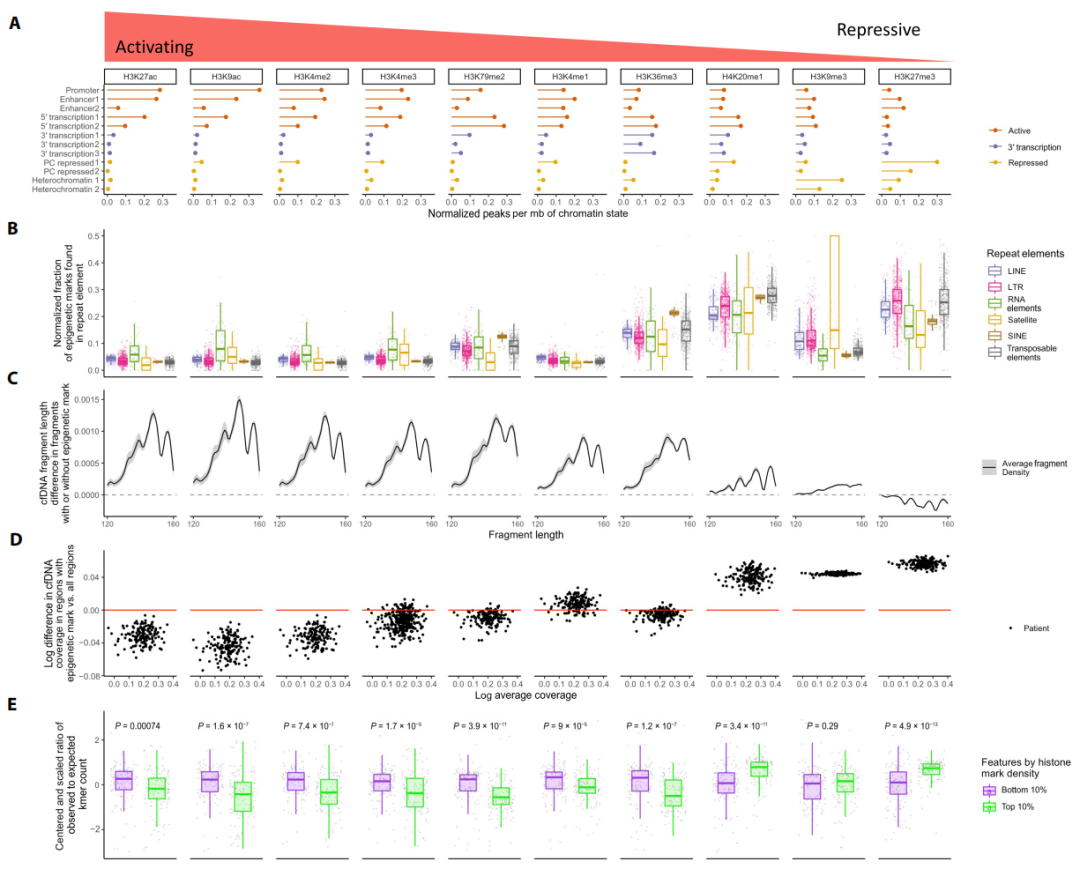
图4. 表观遗传状态对cfDNA中重复序列表达的影响。
研究团队评估了ARTEMIS血液检测能否识别癌症患者体内肿瘤的起源位置,并使用PCAWG参与者的信息进行训练(图5)。结果显示,ARTEMIS能够对肿瘤组织来源进行分类,平均准确率为78%。
最后,研究团队将ARTEMIS和DELFI相结合,对226名患有乳腺癌、卵巢癌、肺癌、结肠癌、胆管癌、胃癌或胰腺癌患者的血液样本进行评估。结果显示,ARTEMIS-DELFI准确地将不同癌症类型的患者分类,平均准确率为68%;当该方法被允许提示两种可能肿瘤类型时,准确率提高至83%。特别地,ARTEMIS和ARTEMIS-DELFI评分与治疗期间观察到的cfDNA突变等位基因分数相关,且ARTEMIS-DELFI评分与无进展生存期相关。
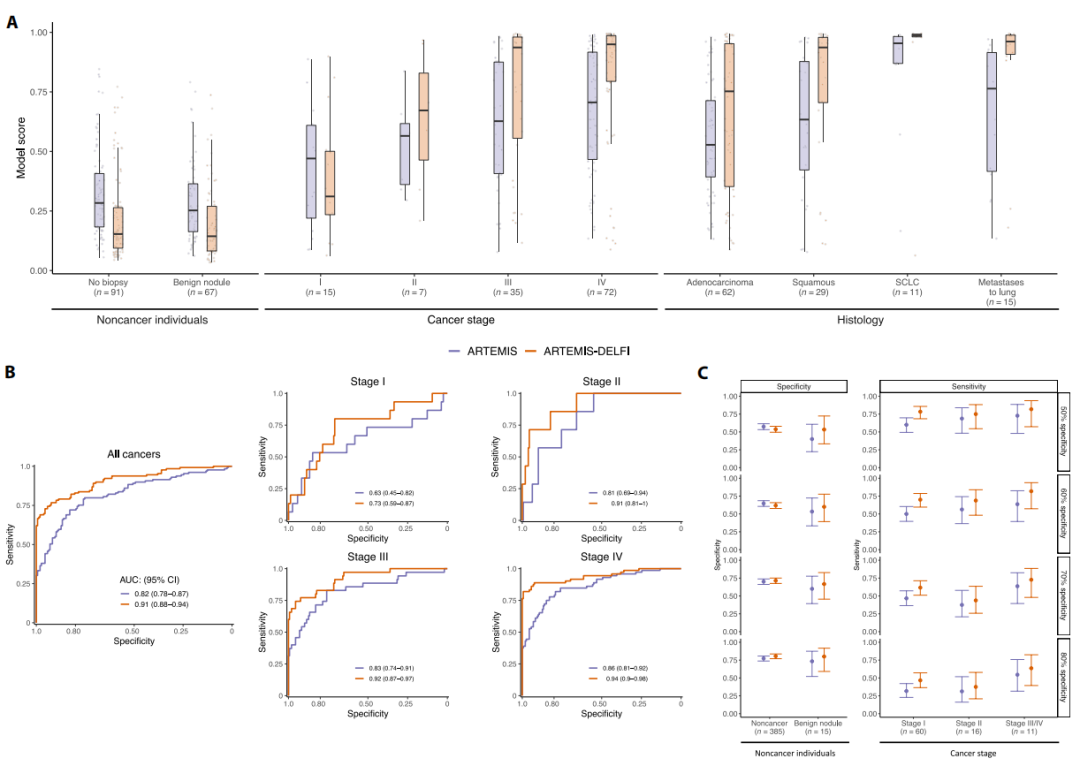
图5. ARTEMIS和ARTEMIS-DELFI使用cfDNA检测肺癌。
综上所述,该研究利用ARTEMIS重建了人类癌症的全基因组重复元件图谱,并反映了癌症的潜在变化。ARTEMIS单独或与其他全基因组特征相结合,可为癌症的无创检测、监测和组织起源确定提供途径。该研究为癌症基因组提供了独特的见解,并证明全基因组kmer重复图谱在癌症检测、表征和监测的组织和血液生物标志物的实用性。
论文原文:
Annapragada AV, Niknafs N, White JR, et al. Genome-wide repeat landscapes in cancer and cell-free DNA. Sci Transl Med. 2024;16(738):eadj9283. doi:10.1126/scitranslmed.adj9283
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)